Сепсис: анализ многомерных данных на пути понимания биологического значения сепсиса и клиническое применение новых знаний
Schuurman et al. Intensive Care Medicine Experimental (2021) 9:27
Обзор
Сепсис: анализ многомерных данных на пути понимания биологического значения сепсиса и клиническое применение новых знаний
Перевод оригинальной статьи «Sepsis: deriving biological meaning and clinical applications from high‑dimensional data»
Авторы: Alex R. Schuurman, Tom D. Y. Reijnders, Robert F. J. Kullberg, Joe M. Butler, Tom van der Poll, W. Joost Wiersinga
Center for Experimental and Molecular Medicine, Amsterdam UMC, Academic Medical Center, University of Amsterdam, Noord-Holland, Amsterdam 1105 AZ, The Netherlands.
Amsterdam Institute for Infection and Immunity, Amsterdam UMC, Noord-Holland, Amsterdam 1105 AZ, The Netherlands.
Введение
Определение сепсиса было обновлено в 2016 году и с этого момента сепсис определяется как угрожающая жизни органная дисфункция, вызванная дисрегуляцией иммунного ответа (ответа хозяина). Это определение охватывает группу пациентов, которая обладает выраженной гетерогенностью как со стороны клинических характеристик, так и со стороны лежащей в основе патофизиологии. Патофизиология сепсиса многогранна и очень сложна для полного ее понимания. Здесь и вовлечение в процесс конкурирующих процессов со стороны иммунной системы в виде чрезмерной ее активации и супрессии, активация системы комплемента, коагулопатия, эндотелиальная дисфункция, разрушение микробиома кишечника, метаболическое ре-программирование клеток иммунной системы [2, 3]. Очень длительное время сепсис остается ведущей причиной смерти во всемирном масштабе, но до настоящего времени терапия сепсиса, основанная на ясных и точных целях, остается невозможной, что делает более полное понимание патофизиологии сепсиса просто настоятельной необходимостью в целях улучшения терапии сепсиса и клинических исходов [4—6].
На помощь приходит анализ многомерных данных - все большее количество исследований используют данный анализ для понимания такого сложного процесса, как сепсис. Прогресс в технологиях увеличивает возможности для «-омик» измерений всех составляющих молекулярного слоя - РНК, белков или метаболитов, что обеспечивает беспристрастный взгляд в текущие процессы заболевания. Три основных цели, на которые направлены новые методологии клинического изучения сепсиса - иммунный ответ (ответ хозяина), развитие диагностики и обнаружение клинически значимых кластеров. Однако, мы все еще испытываем значительные трудности на пути создания всеобъемлющей модели механизмов патофизиологии сепсиса.
В этой работе мы подводим предварительные итоги исследований, в основе которых лежит анализ многомерных данных в такой области исследований, как «-омики», отдавая тем самым дань центральной догме молекулярной биологии: геномика, эпигеномика, транскриптомика, протеомика, липидомика и микробиомика. Данные считаются многомерными тогда, когда количество измеренных характеристик превышает количество взятых проб/образцов (англ. samples). Задача нашей работы – привлечь внимание к исследованиям, которые представляют собой важные достижения в данной области или иллюстрируют конкретную тему. Важное замечание - наш нарративный (повествовательный) обзор не является исчерпывающим обзором литературы. Мы сосредоточились на тех областях, что могут дать нам новые знания о сепсисе, выделяя при этом текущие тренды и будущие вызовы (Таб. 1). Полномасштабные клинические исследования не включены в наш обзор, мы больше сосредоточились на исследованиях биологических образцов человека. В конце обзора мы исследовали потенциальные и существующие проблемы на пути интеграции данных, включающих в себя несколько «-омиков».
Геномика
Область знаний для геномики сосредоточена на структуре, катрировании, кодировании геномов и определения их функции. Одной из основных целей геномики является идентификация генетических модификаций в геноме человека, что вызывают риски развития болезней. Идентификации можно достичь путем изучения ассоциаций на уровне всего генома (англ. genome wide association study или GWAS), при котором миллионы одно-нуклеотидных полиморфизмов (англ. single-nucleotide polymorphisms или SNPs) во всем геноме измеряются в дизайне «случай-контроль». Каждый субъект генотипируется с помощью чипа анализатора SNP, который напрямую измеряет до 1 миллиона модификаций во всем геноме, и это количество измерений можно увеличить (до 90 миллионов) [7].
Общеизвестный факт - инфекционные заболевания во всем мире определяют высокую смертность среди детей и подростков (до и во время репродуктивного возраста), что делает инфекционные патогены одной из самых сильных сил эволюционного отбора в человеческой популяции. Более того, постулируется, что инфекционные патогены сформировали геном человека таким образом, что увеличилась частота аллелей защитных модификаций в генах, связанных с иммунитетом. Связь между генетикой и выживаемостью от инфекционных заболеваний была доказана в важнейшем исследовании Sørensen and colleagues, которые сообщили, что у взрослого человека риск смерти от инфекции увеличивается более чем в пять раз в случае, если один из его биологических родителей умер от инфекции в возрасте до 50 лет [9]. Примечательно, что этот риск превышает риск смерти от злокачественного новообразования или от сердечно-сосудистого заболевания. Обнаружение генетических модификаций, связанных с летальностью/выживаемостью от инфекционных болезней, и более глубокое понимание соответствующих физиологических механизмов открывают нам многообещающие возможности в разработке новых методов лечения сепсиса.
На сегодняшний день исследования сепсиса с использованием GWA в основном фокусировались на таких клинических исходах, как 28-дневная летальность [10, 11]. Такой дизайн исследования подразумевает наличие групп вмешательства и контроля, пациенты в которых имеют сепсис, а статус «случай/контроль» определяется летальностью. Проведенные исследования выявили SNP в таких генах, как FER, но точные механизмы, посредством которых полиморфизмы проявляют свой защитный или вредный эффект, еще предстоит выяснить. К примеру, наличие s4957796 SNP в гене FER существенно увеличивает выживаемость пациентов с сепсисом, возможно за счет роли FER в регуляции адгезии клеток, рекрутмента лейкоцитов и дисфункции интестинального барьера [10]. Но здесь важно отметить, что модификации гена FER ассоциируются с выживаемостью при сепсисе, причиной которого стала пневмония, но не ассоциируются с летальностью в более гетерогенной когорте пациентов с сепсисом, причиной которого стали абдоминальные инфекции или пневмония, что может свидетельствовать в пользу различий механизмов и их зависимости от источника инфекции [11].
В дополнение к выявлению модификаций, что оказывают влияние на выживаемость при сепсисе, следует уделить внимание и модификациям, что оказывают влияние на риск развития сепсиса. Мета-анализ исследований генов-кандидатов (целеполагание на подозрительные гены) сообщил о ряде SNP в генах, кодирующих рецепторы распознавания (англ. pattern recognition receptors) и цитокины, что значительно ассоциировалось с риском развития сепсиса, включая сюда TLR4rs4986790 и TNFArs1800629, которые были изучены в 25 исследованиях [12]. Если принять во внимание, что Toll-like рецепторы (TLRs) являются чрезвычайно важными для врожденной иммунной системы, а фактор некроза опухоли альфа (TNF-α) является важнейшим для провоспалительных цитокинов, то генетические модификации в генах, ответственных за них, потенциально оказывают сильное влияние на иммунный ответ (ответ хозяина) во время инфекции. Но все же остается неизвестным, действительно ли такие полиморфизмы опосредуют риск развития сепсиса. Исследования GWA, дизайн которых предполагал бы наличие двух групп пациентов, одна из которых пациенты с сепсисом, а вторая - пациенты из общей популяции, возможно и ответили бы на этот вопрос, но насколько мы знаем, таких исследований еще не проводилось.
Эпигеномика
Один из основных путей регуляции транскрипции генов - эпигенетические изменения в виде метилирования ДНК и модификации гистонов. К примеру, метилирование ДНК в промоторной области гена подавляет транскрипцию гена [13]. По шкале «омиков» этот процесс получил название эпигеномики. В контексте сепсиса недавние исследования стали показывать роль, что эпигенетика играет в патогенезе сепсиса. Binnie and colleagues выполнили анализ метилирования ДНК по всему эпигеному образцов цельной крови, взятых у 68 пациентов с сепсисом и у 66 пациентов (все взрослые), находящихся в критическом состоянии по другим, отличным от сепсиса, причинам [14]. Они определили 668 различающихся метилированных областей (англ. differentially methylated regions или DMRs), большинство из которых были гиперметилированны. После анализа DMR-содержащих генов было показано, что пути, связанные с противовоспалительным ответом и иммунным ответом Т-хелперов 1 типа, были обогащены гиперметилированными генами. И наоборот, пути, включающие в себя отрицательную регуляцию продукции IFNγ были обогащены гипометилированными генами. И наконец, авторы нашли определенные наборы (сеты) метилированных генов, которые коррелировали с необходимостью в проведении вазопрессорной терапии и тяжестью состояния, таким образом предполагается их связь с клиническими характеристиками. В другом исследовании изучалось глобальное метилирование ДНК моноцитов у пациентов с сепсисом [15]. Сепсис ассоциировался с изменениями метилирования генов, релевантных к функции моноцитов, включая тех, что вовлечены в воспаление, опосредованное хемокин-цитокиновым сигнальным путем (гиперметилировние), и главного комплекса гистосовместимости II класса (гипометилирование), и все это поддерживает роль метилирования ДНК в регуляции экспрессии генов при сепсисе.
Нельзя не упомянуть еще об одном механизме, известном как регуляция транскриптома, хотя это и не относится к эпигеномике, в виде использования некодирующих РНК, как длинно-цепочечных, так и микроРНК. Этот механизм недавно был изучен при сепсисе, используя новую генерацию микроанализаторов, на лейкоцитах. Было найдено, что длинная некодирующая ДНК и, в меньшей степени, короткая некодирующая ДНК при сепсисе были значительно изменены по сравнению со здоровым состоянием [16]. Будущие механистические исследования сепсиса должны быть нацелены на интеграцию эпигеномики и транскриптомики для того, чтобы определить, насколько вариативность транскриптомики зависит от метилирования, модификации гистонов и некодирующих транскриптов.
Транскриптомика
Широкий спектр молекул РНК, транскрибируемых из генома, выполняет разнообразные функции в виде продукции белков и регуляции экспрессии генов. Большинство исследований транскриптомики при сепсисе изучало мессенджер (информационная) РНК (мРНК) — экспрессию генов, что приводит к продукции соответствующего белка —но в дальнейшем фокус расширился до регулирующих некодирующих форм, таких как микроРНК и длинная некодирующая РНК. При измерении транскриптома микроанализаторы позволяют определять большое количество предопределенных генов в то время, как новейшая генерация секвенирования РНК определяет все присутствующие молекулы РНК, включая новейшие и альтернативно сплайсированные транскрипты [17].
Огромные объемы данных, полученных с помощью транскриптомики, способствовали разработке новейших диагностических тестов/анализаторов для сепсиса. В то время как секвенирование всех РНК в биологических пробах является весьма затратным методом, как со стороны расходов, так и со стороны требуемого времени для получения окончательного результата, комбинация генов, диагностическая ценность которых подтверждена, может послужить основой быстрого и относительно недорого, частично автоматизированного ПЦР теста. К примеру, несколько наборов генов, полученных из лейкоцитов цельной крови, валидированы для дифференциации системного воспаления, вызванного инфекцией, от стерильного воспаления [18]. Такие наборы генов включают в себя тесты Sepsis MetaScore [19], SeptiCyte ™ LAB [20] и the FAIM3:PLAC8 ratio [21]. Такие диагностические тесты в сочетании с другими тестами способны улучшить общую прогностическую ценность, а в отделениях интенсивной терапии, теоретически, эти тесты могут оказать влияние на сокращение применения антибиотиков, что позволит избежать вредных побочных эффектов и развития резистентности к антиомикробным препаратам, и даже побудить врачей рассматривать альтернативные не-инфекционные диагнозы.
Данные транскриптомики могут быть использованы для так называемой кластеризации пациентов с сепсисом или для формирования подгрупп, каждая из которых имеет присущие ей значимые клинические характеристики и которые уже получили свое название «эндотипы» пациентов. При условии постоянства таких эндотипов, быстрая их идентификация соотносится с требованиями прецизионной (точной) медицины и, применительно к сепсису, принятие решения о лечении может базироваться на специфических биологических процессах, лежащих в основе заболевания, а не на таком абсолютно неспецифическом определении, как «подозрение на инфекцию с органной дисфункцией» [18]. За последние годы было охарактеризовано уже несколько эндотипов: эндотипы А и В в педиатрическом сепсисе [22], сигнатуры ответа на сепсис (англ. sepsis response signature или SRS) 1 и 2 [23], Mars 1–4 [24], а также инфламопатические (при нарушениях воспалительного ответа), адаптивные и коагулопатические эндотипы [25]. На молекулярном и патофизиологическом уровне некоторые из этих эндотипов пересекаются. К примеру, SRS2, Mars3 и адаптивный эндотипы описывают одну и ту же группу пациентов с чрезмерной регуляцией генов адаптивной иммунной системы (ассоциируется с низкой летальностью) [24, 25]. Эти эндотипы теоретически могут «обогатить» прогноз, ведь ранняя идентификация пациентов с определенными патофизиологическими характеристиками приведет к большей вероятности положительного ответа на определенную терапию. Но для этого требуются ответы на множество вопросов, таких как постоянство/стабильность эндотипов с течением времени, обобщаемость результатов и практическая их реализация [18, 26]. Исследований, которые бы переводили бы эти эндотипы в клиническую практику, невероятно мало и это при том, что уже описаны различные ответы на терапию. Сюда необходимо отнести исследование VANISH, где введение гидрокортизона ассоциировалось с увеличением летальности у эндотипа SRS2 [27] и это при том, что исследование педиатрического сепсиса показало, что эндотип В, характеризующийся высоким уровнем экспрессии глюкокортикоидных рецепторов, при назначении лечения глюкокортикоидами показывает улучшение клинических исходов [28].
Оценка экспрессии генов в лейкоцитах, содержащихся в цельной крови, или в специфических фракциях клеток, может быть использовано при изучении иммунного ответа при сепсисе. К примеру, Claushuis et al.[29] стратифицировали пациентов с сепсисом, находящихся в критическом состоянии, основываясь на количестве тромбоцитов и нашли, что даже после корректировки на тяжесть состояния и на другие вмешивающиеся факторы пациенты с наиболее выраженной тромбоцитопенией показывали большие нарушения в иммунном ответе (например, в виде чрезмерной экспрессии генов, связанных с системой комплемента) по сравнению с пациентами, у которых сохранялось нормальное количество тромбоцитов. Используя доступные (публичные) данные, Zador et al. [30] показали существенное дублирование путях экспрессии генов, вовлеченных в ответ на легочный сепсис, абдоминальный сепсис и травму. Используя CIBERSORT, метод получения фракций популяции лейкоцитов на основании данных о совокупной РНК [31], они нашли более высокую частоту летальности в группе пациентов, которая характеризовалась меньшим количеством циркулирующих нейтрофилов.
Недавно появившийся и очень многообещающий метод в области транскриптомики сепсиса основан на секвенировании РНК отдельных клеток, которые разделяются на отдельные капли, после чего комбинируются с уникальными РНК-шариками со штрих-кодом (англ. unique RNA-barcoded beads), что позволяет идентифицировать транскриптом каждой клетки [32]. Подобные технологии генерируют огромные массивы данных по каждому субъекту исследования, но высокие финансовые затраты препятствуют получению больших размеров выборки в массовых исследованиях транскриптома. Все это ограничивает как мощность сравнения между субъектами, так и воспроизводимость (генерализацию) результатов. Но есть и преимущества, одним из которых является возможность РНК-секвенирования отдельной клетки идентифицировать новые типы клеток и состояний, что утеряны в больших массивах данных. Reyes et al. [33] провели РНК-секвенирование мононуклеарных клеток периферической крови в когорте из 29 пациентов с сепсисом (основная причина - инфекция мочевыводящего тракта) и нашли новое состояние моноцита (названное “MS1”), которое отсутствовало у здорового контроля и у пациентов с инфекцией мочевыводящего тракта, но без сепсиса. Важно, что авторам удалось оценить надежность такого MS1 состояния несколькими путями. Они валидировали траскриптомную MS1сигнатуру во внешних данных о групповой последовательности РНК, им удалось получить клетки с подобными MS1 характеристиками путем стимуляции прекурсорных клеток костного мозга липополисахаридом (ЛПС), а плюсом ко всему стало определение маркеров поверхности, которые позволяют клеткам переходить в MS1 состояние. Другое исследование, изучавшее семь пациентов с сепсисом и четырех здоровых волонтеров (группа контроля), подтвердило ранее полученные данные об изменениях на уровне одной клетки, такие как подавление регуляции генов, связанных с HLA-DR, и нарушения метаболизма энергии в кластерах моноцитов [34]. В третьем исследовании, что включило в себя семь пациентов с сепсисом — у трех из них развился острый респираторный дистресс-синдром, — была ясно показана стимуляция регуляции генов, связанных с сигнальным путем интерферона I типа при ОРДС, потенциально это может быть обусловлено отсутствием SOCS3 (отрицательный регулятор сигнальных путей цитокинов) во всем кластере моноцитов у данной группы пациентов [35].
Протеомика
Протеомика в иерархии «-омиков» часто рассматривается, как шаг вниз, оставляющий сверху геномику и транскриптомику. Протеомика направлена на анализ белков, которые производятся или модифицируются организмом, и в первую очередь этот анализ касается состава, активности и структуры. Измерения проводятся посредством электрофореза в геле, иммуноанализа антител и жидкостной хроматографией с последующей масс-спектрометрией [36].
Белки состоят из аминокислот, которые были собраны на рибосоме на основе генетической информации, предоставляемой матричной РНК (мРНК), а сам процесс называется трансляцией. После рибосомной трансляции белки часто изменяются путем добавления или удаления определенных молекул (таких как фосфатные или метильные группы), что может заметно изменить функцию белка. Посттрансляционные модификации делают протеом очень динамичным и скрывают его прямую связь с геномом и транскриптомом.
Белки в качестве конечного продукта транскрипции, трансляции и модификации могут предоставить механистическое понимание и/или послужить биомаркерами. Несколько исследовательских групп уже провели анализы протеома при сепсисе [37, 38]. Например, De Coux et al. [39] сравнили выживших и не выживших пациентов с сепсисом в небольшой когорте пациентов отделений неотложной помощи и нашли более 90 белков плазмы, что эксклюзивно наблюдались только в одной из двух групп. Используя Киотскую энциклопедию генов и геномов (англ. the Kyoto Encyclopedia of Genes and Genomes), авторы определили специфически стимулированные пути (каскады), такие как внешний коагуляционный каскад и каскад комплемента. Другие исследователи сообщили об изменениях протеома в связи с возрастом в популяции из 19 пациентов старческого возраста с сепсисом [40].
В более обширном и многостадийном исследовании Langley et al. [41] нацелились и на метаболом, и на протеом плазмы пациентов с сепсисом сразу после их поступления в отделение неотложной помощи и после 24 часов госпитализации. Дизайн исследования подразумевал наличие трех групп пациентов с сепсисом или с синдромом системного воспалительного ответа (ССВО) — группа исследования (150 пациентов), группа валидации 1 (52 пациента) и группа валидации 2 (90 пациентов). Путем перекрестной корреляции и иерархической кластеризации протеома и метаболома авторы дали характеристику известным метаболическим реакциям, открывая анализируемые вещества и пути, такие как глубокий дефект бета-окисления жирных кислот, которые отличают выживших от не выживших. Интересно, что в этом исследовании ни метаболом плазмы, ни протеом плазмы не позволили дифференцировать тяжелый сепсис от септического шока у выживших пациентов. Об изменениях в пути метаболизма жиров было сообщено и в другом исследовании, в котором проводилось сравнение плазмы между 23 здоровыми волонтерами, 20 выжившими от сепсиса и 13 не выжившими от сепсиса на момент поступления в госпиталь и на исходе 7 суток [42]. Подавление регуляции аполипротеинов и нарушения в пути метаболизма холестерола отличают пациентов с сепсисом от здорового контроля в то время, как дисрегуляция пути актина цитоскелета была больше выражена у не выживших от сепсиса пациентов, чем у выживших.
Несмотря на то, что эти исследования подчеркивают потенциал протеомных данных, в них также указывается и на существенные ограничения измерений протеома в плазме. Сложно сделать механистические выводы из различающихся сигнатур в таких образцах, как плазма или моча [43], поскольку происхождение аналитов часто остается неопределенным. Эту проблему можно решить, сосредоточив внимание на более конкретном образце, таком как тромбоциты или нейтрофилы [44, 45], или пойти путем проверки результатов in vitro.
Липидомика и метаболомика
Жиры есть ничто иное, как необходимое условие существования клеток. Жиры несут в себе важнейшие структурные и биоактивные функции. Жиры являются блоками для строительства мембран клеток. Как биоактивные молекулы, жиры играют ключевые роли во многих клеточных процессах, таких как метаболизм энергии в клетке, транспорт медиаторов и обеспечение сигнальных путей между клетками (англ. cell–cell signaling) [46]. И в таком контексте изучение метаболитов (метаболомика) тесно связано с липидомикой просто потому, что для их измерения применяются одинаковые методы, в основном это хроматография с последующей масс-спектрометрией.
В человеческом теле присутствует более десяти тысяч жировых частиц, которые могут быть разделены на восемь основных классов и намного большее количество подклассов, согласно химической структуре и характеристикам [47].
Такая вариабельность жиров, широкий диапазон концентраций жиров в биологических жидкостях и ограничений, присущих предыдущим инструментам детекции жиров, наше понимание липидома отстает от понимания генома и транскриптома.
Большинство исследований жиров при сепсисе было сосредоточено на потенциальной роли специфических жиров как биомаркеров плазмы. К примеру, уровень холестерола липопротеинов высокой плотности (англ. high-density lipoprotein cholesterol или HDL-C) при сепсисе снижается, что может ассоциироваться с неблагоприятными клиническими исходами [48]. HDL-C может связывать и изолировать жиры, полученные из патогенов и потенциально несущие вред, что привело к гипотезе о защитной роли HDL-C при бактериальной инфекции [49]. Mecatti et al. [50] измерили часть липидома в плазме у 21 пациента с ССВО и у 21 пациента с сепсисом. Множество жировых частиц, таких как глицеросфинголипиды и простагландины, в большем количестве наблюдались у пациентов с сепсисом в то время, как L-octanoylcarnitine показал свои возможности, как прогностический фактор, помогая отличать выживших пациентов от не выживших. В еще одном исследовании изучались связи жировых сигнатур с ответом на терапию у 21 пациента с септическим шоком. Исследование показало, что уровень лизофосфатилилхолина повышается только у пациентов, отвечающих на проводимую терапию [51].
Эйкозаноиды возможно являются наиболее изучаемыми жирами в контексте инфекции. Грубо их можно разделить на провоспалительные медиаторы и на медиаторы, способствующие разрешению процесса. Продуцируются они из полиненасыщенных жирных кислот в лейкоцитах [52]. Нестероидные противовоспалительные лекарственные средства используются на протяжении десятилетий для подавления провоспалительных эйкозаноидов (таких как простагландин). В то же время недавно было обнаружено, что эйкозаноиды могут активно способствовать разрешению воспаления. На животных моделях сепсиса было показано, что вмешательства с такими медиаторами разрешения воспаления, как резолвины (англ. resolvins) и протектины (англ. protectins), увеличивают поглощение фагоцитами, что способствует увеличению бактериального клиренса, снижая при этом потребность в антибиотиках [53]. Эйкозаноиды могут послужить и биомаркерами — в исследовании Dalli et al. [54] было показано, что уровень жиров-медиаторов в плазме критически больных пациентов с сепсисом коррелировал с летальностью и развитием ОРДС.
Жиры и метаболиты имеют большое значение в обеспечении нормальной функции клетки. Следовательно, измерение метаболома и липидома в изолированных клетках иммунной системы при инфекции может принести пользу в виде информации о нарушениях в жизнедеятельности клетки. К примеру, все большее количество исследований на эту тему показывает, что изменения путей метаболизма энергии, также называемое метаболическим ре-программированием, может нарушать функциональность иммунных клеток [55]. Для иллюстрации можно привести пример, когда фенотипами макрофагов, ранжируемых от про-воспалительных до анти-воспалительных, можно управлять, контролируя перекисное окисление жирных кислот (англ. fatty acid oxidation) [56]. Khalic et al. [57] обнаружили в метаболических сигнатурах сыворотки крови 33 пациентов, находящихся в критическом состоянии, что как повышение продуктов бета-окисления жирных кислот в митохондриях, так и их снижение, было связано с летальностью и выдвинули гипотезу о наличии так называемого «коридора безопасности», или определенного диапазона, в котором должен поддерживаться клеточный метаболизм жирных кислот.
Понимание роли жиров и метаболитов может дать не только ценную информацию о внутреннем состоянии и о структурных изменениях клеток иммунной системы, но и пролить свет на взаимодействие между хозяином и патогеном (англ.host–pathogen interactions) [58, 59], что все вместе, возможно, проложит дорогу к поиску новых и эффективных вмешательств. В совокупности все исследования указывают на то, что нарушения в липидоме и метаболоме могут играть значительную роль в иммунном ответе при сепсисе, но все же эта область наших знаний в настоящее время находится в фазе изучения.
Микробиомика
Микробиом человека содержит триллионы микробов, что колонизировали человеческое тело, а самое большее их количество содержится в кишечнике [60]. Развитие культуро-независимых технологий, таких как метагеномное секвенирование 16S рРНК и метод «дробовика», позволяет исследовать микробные сообщества и их роль в здоровье и при заболеваемости. Множество экспериментальных и эпидемиологических исследований уже показали важную, правда до конца не понятную, роль микробиома кишечника при сепсисе [61]. Нарушения микробиома при госпитализации по причине применения антибиотиков могут привести к повторным госпитализациям, но уже причиной госпитализации может стать сепсис [62]. Несколько проспективных исследований, в которых изучались изменения микробиома у пациентов, находящихся в критическом состоянии, показали снижение облигатных аэробов и чрезмерный рост потенциально патогенных бактерий, таких как Staphylococcus and Pseudomonas spp.[63–66]. Здесь следует заметить, что такие результаты могут быть обусловлены еще и вмешивающимися факторами в виде антибактериальной терапии или других видов терапии, таких как энтеральное/парентеральное питание, применение ингибиторов желудочной секреции и седативных препаратов [61].
Нарушения микробиома не ограничивается только лишь микробиомом кишечника. Как недавно было показано, нарушение микробиома легких при критических состояниях приводит к колонизации дыхательных путей бактериями, больше ассоциированными с кишечником [67]. На животных моделях сепсиса (мыши) бактерии, обнаруживаемые в легких, скорее всего происходили из нижних отделов желудочно-кишечного тракта (ЖКТ), а у пациентов с ОРДС высокий уровень специфичных для ЖКТ бактерий в бронхоальвеолярной жидкости ассоциируется с тяжестью заболевания [68]. Более того, недавнее исследование показало наличие связи между снижением разнообразия легочного микробиома с увеличением летальности у пациентов с внелегочным сепсисом [69].
Методы для измерения микробиома продолжают эволюционировать. Амплификация и секвенирование генов-маркеров сейчас становится практически основным методом, чему способствует еще и его эффективность в контексте затрат, при котором возможно получить обзор целого сообщества микроорганизмов одного типа (16S рРНК для бактерии), но и его недостаточно для решения задач в этой области. Для полной картины исследователям необходимо учитывать все сообщества микроорганизмов, что могут быть представлены в одном образце с последующей интеграцией в бактериом, виром, фунгиом и протозом [70]. Метагеномическое секвенирование измеряет все гены в образце микробиома, включая сюда вирусные ДНК и ДНК эукариот, тем самым обеспечивая высокое таксономическое разрешение и выводы о функциональных возможностях [71]. Применимые сейчас методы измерения метагенома используют так называемое секвенирование «короткого чтения» (англ. short-read sequencing), что позволяет одновременно секвенировать смесь микробных генов, но результаты здесь можно отнести к суб-оптимальным потому, что такое короткое чтение может не учитывать наложение множества микробных геномов [72]. Решить эту проблему можно с помощью секвенирования «долгого чтения» (англ. long-read sequencing), что может способствовать получению более надежных результатов.
Несмотря на то, что сейчас есть возможность определить состав микробиома достаточно подробно, текущие терапевтические вмешательства, такие как пробиотики или селективная деконтаминация, часто не специфичны и используют подход, известный как «один-размер-подходит-для-всех». Совершенствование наших знаний микробиома при здоровье и при болезнях, особенно при сепсисе, приведет к развитию персонализированной, основанной на микробиоме, терапии [73].
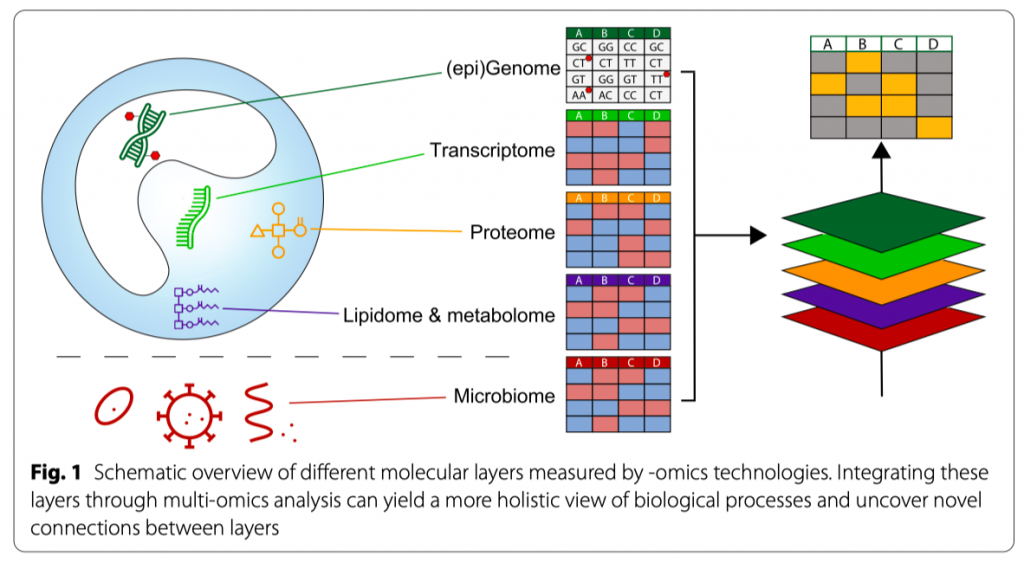
Интеграция мульти-омических данных
Изречение поэта и эссеиста Хорхе Луиса Борхеса: - «Все касается всего». Все молекулярные слои, о которых мы рассуждали в предыдущих главах, взаимосвязаны и постоянно оказывают влияние друг на друга (Рис. 1). Интеграция этих слоев молекул может дать более целостный взгляд на биологические процессы и выявить новые, доселе неизвестные, связи между слоями. Достижения в области биоинформатики подарили нам возможность проводить интегрированный и конкурентный анализ двух и более «-омик» слоев (мульти-омик), но в области изучения сепсиса таких исследований было очень мало [74, 75]. Первое исследование интеграции геномики и транскриптомики при сепсисе было проведено Davenport and colleagues [23], которые предположили, что «индивидуальная гетерогенность транскриптомного ответа на сепсис может модулироваться генетическими вариациями». Они изучили экспрессию генов в локусах количественных признаков (англ. Quantitative Trait Loci или eQTLs), для которых генотип в значительной мере связан с экспрессией генов. В когорте с сепсисом было обнаружено, что гены цис-регуляторных элементов eQTL (где SNP и ген локально совпадают на хромосоме) обогащены путями, имеющими отношение к сепсису, такими как вирусная респираторная инфекция и клеточный иммунный ответ. Гены цис-eQTL включали TLR4 и TNF, что свидетельствует о том, что у пациентов с сепсисом на экспрессию лейкоцитарных генов ключевых иммунных регуляторов действительно влияет генетика.
Мульти-омики могут также улучшить диагностику путем увеличения соразмерности данных. Wong et al. [76] разработали классификатор, в котором сочетались транскриптомные маркеры и белки сыворотки крови, и этот новый классификатор мог помочь провести различие между выжившими и не выжившими детьми с септическим шоком, что превосходило возможности более раннего классификатора, основанного только на белках сыворотки крови. Сочетание геномики с измерениями метаболитов и цитокинов выявило «путь спасения» метионина, как регулятора сепсиса, что также может служить предиктором прогноза у пациентов [77]. Как было показано ранее, Langley et al.[41] смогли провести дифференциацию между выжившими и не выжившими пациентами с сепсисом, используя для этого интегрированный, протеомик и метаболомик, подход.
Методы, основанные на мульти-омике, все еще находятся в зачаточном состоянии, а их дальнейшее применение требует решения нескольких проблем. Во-первых, большинство инструментов мульти-омики требуют нормализации данных, фильтрации шума и других подготовительных шагов. Без стандартизации подходов становится все труднее сравнивать полученные в разных исследованиях результаты. Во-вторых, каждый «-омик» действует в своей временной шкале, что делает трудным проводить измерения в едином временном стандарте для определения причинно-следственных связей между слоями. В-третьих, до настоящего момента не имеется согласия в выборе того необходимого (правильного) анализа и требуемая для этого комбинация «-омиков».
Subramanian and colleagues предложили классифицировать инструменты по технике (например, байесовские, сетевые методы или факторизация) и по тем биологическим вопросам, которые они могут решить (например, определение подгрупп, открытие биомаркеров или получение информации о патофизиологии) [75]. Такие основанные на консенсусе рекомендации могут быть полезны исследователям при выборе такого аналитического метода, который лучше всего соответствует вопросам их исследования
Обсуждение
Редукционизм - вера в то, что каждый процесс в природе может быть разбит на его составные части и объяснен ими. Мы знаем, что клетки не двумерные, что пути не похожи на прямые стрелки, что множество реакций происходит только после достижения некоторого критического (дис) баланса, часто с нелинейными последствиями. Тем не менее, тестирование и визуализация процессов описанными выше способами помогает нам анализировать (можно сказать препарировать) такие сложные проблемы, как сепсис. Вся наука в той или иной мере редукционизм, а технологии, основанные на «-омиках», позволяют получить всеобъемлющее представление об исследуемых процессах. Такие исследования сулят знаковые открытия, выходящие за пределы нашего сегодняшнего понимания. Но и здесь, для подтверждения и интерпретации результатов, полученных после интеграции и анализа многомерных данных, мы полагаемся на результаты традиционных экспериментальных исследований.
Несмотря на все более изощренные методы анализа, измерение одного субстрата в один момент времени вряд ли предоставит нам полную, или исчерпывающую, информацию о биологическом процессе. Пертурбации при сепсисе совсем не обязательно ограничиваются специфическим компартментом, будь то бронхоальвеолярное пространство, периферическая кровь или что-то еще, а являются частью общесистемных и взаимосвязанных изменений. Мы знаем, что процессы с течением времени изменяются, и мы видим это на примере прогрессирования сепсиса, что приводит к временной неоднородности. Отсюда и требования к исследованиям, которые должны включать в себя продольные измерения множества биологических компартментов. И здесь важно отметить, что даже самые сложные и изощренные измерения с последующим биоинформационным анализом не способны компенсировать небрежно собранные образцы и/или предвзятый дизайн исследования.
Касательно транскрипотомики - здесь уже принята обязательная практика в виде обмена необработанными данными, что привело к плодотворному повторному использованию данных, полученных в одной или нескольких когортах, и позволяет проводить мета-анализы множества наборов данных. Мета-анализы увеличивают мощность исследований и их эффективность, что помогает в обнаружении и консолидации паттернов данных с потенциальной клинической значимостью, как например, различные эндотипы при сепсисе. Несмотря на то, что большие массивы данных снижают случайную ошибку, они не могут предотвратить предвзятость/смещение (bias) и риск предвзятости особенно высок тогда, когда человек не осознает всех сложностей и хитросплетений набора данных и вмешивающих факторов. Мета-анализы больших массивов данных должны принять методологическую строгость, характерную для традиционных клинических эпидемиологических исследований, применяемую, например в Кохрейновском сотрудничестве (англ. the Cochrane collaboration), не исключая при этом тесное сотрудничество с исследователями, что публикуют наборы данных на уровне отдельных пациентов.
Выводы
За последние годы в исследованиях сепсиса был достигнут огромный прогресс, но остается решить еще несколько важнейших задач. Подход, основанный на мульти-омиках, когда сочетаются данные о молекулярных слоях с данными о состоянии биологических компартментов, позволит нам лучше понять патофизиологию сепсиса, что откроет нам путь для развития быстрой диагностики и последующей персонализации терапии сепсиса.

References
1. Singer M, Deutschman CS, Seymour C, Shankar-Hari M, Annane D, Bauer M, Bellomo R, Bernard GR, Chiche JD, Coopersmith CM, Hotchkiss RS, Levy MM, Marshall JC, Martin GS, Opal SM, Rubenfeld GD, Der PT, Vincent JL, Angus DC (2016) The third international consensus definitions for sepsis and septic shock (sepsis-3). JAMA 315:801–810. https://doi.org/10.1001/jama.2016.0287
2. Hotchkiss RS, Moldawer LL, Opal SM, Reinhart K, Turnbull IR, Vincent JL (2016) Sepsis and septic shock. Nat Rev Dis Prim 2:1–21. https://doi.org/10.1038/nrdp.2016.45
3. Van Der Poll T, Van De Veerdonk FL, Scicluna BP, Netea MG (2017) The immunopathology of sepsis and potential therapeutic targets. Nat Rev Immunol 17:407–420. https://doi.org/10.1038/nri.2017.36
4. Rudd KE, Johnson SC, Agesa KM, Shackelford KA, Tsoi D, Kievlan DR, Colombara DV, Ikuta KS, Kissoon N, Finfer S, Fleischmann-Struzek C, Machado FR, Reinhart KK, Rowan K, Seymour CW, Watson RS, West TE, Marinho F, Hay SI, Lozano R, Lopez AD, Angus DC, Murray CJL, Naghavi M (2020) Global, regional, and national sepsis incidence and mortality, 1990–2017: analysis for the Global Burden of Disease Study. Lancet 395:200–211. https://doi.org/10.1016/ S0140-6736(19)32989-7
5. Cavaillon J, Singer M, Skirecki T (2020) Sepsis therapies: learning from 30 years of failure of translational research to propose new leads. EMBO Mol Med 12:e10128. https://doi.org/10.15252/emmm.201810128
6. Rubio I, Osuchowski MF, Shankar-Hari M, Skirecki T, Winkler MS, Lachmann G, La Rosée P, Monneret G, Venet F, Bauer M, Brunkhorst FM, Kox M, Cavaillon JM, Uhle F, Weigand MA, Flohé SB, Wiersinga WJ, Martin-Fernandez M, Almansa R, Martin-Loeches I, Torres A, Giamarellos-Bourboulis EJ, Girardis M, Cossarizza A, Netea MG, van der Poll T, Scherag A, Meisel C, Schefold JC, Bermejo-Martín JF (2019) Current gaps in sepsis immunology: new opportunities for translational research. Lancet Infect Dis 19:e422–e436. https://doi.org/10.1016/S1473-3099(19)30567-5
7. Marchini J, Howie B, Myers S, McVean G, Donnelly P (2007) A new multipoint method for genome-wide association studies by imputation of genotypes. Nat Genet 39:906–913. https://doi.org/10.1038/ng2088
8. Karlsson EK, Kwiatkowski DP, Sabeti PC (2014) Natural selection and infectious disease in human populations. Nat Rev Genet 15:379–393. https://doi.org/10.1038/nrg3734
9. Sørensen TIA, Nielsen GG, Andersen PK, Teasdale TW (1988) Genetic and environmental influences on premature death in adult adoptees. N Engl J Med 318:727–732. https://doi.org/10.1056/nejm198803243181202
10. Rautanen A, Mills TC, Gordon AC, Hutton P, Steffens M, Nuamah R, Chiche JD, Parks T, Chapman SJ, Davenport EE, Elliott KS, Bion J, Lichtner P, Meitinger T, Wienker TF, Caulfield MJ, Mein C, Bloos F, Bobek I, Cotogni P, Sramek V, Sarapuu S, Kobilay M, Ranieri VM, Rello J, Sirgo G, Weiss YG, Russwurm S, Schneider EM, Reinhart K, Holloway PAH, Knight JC, Garrard CS, Russell JA, Walley KR, Stüber F, Hill AVS, Hinds CJ (2015) Genome-wide association study of survival from sepsis due to pneumonia: An observational cohort study. Lancet Respir Med 3:53–60. https://doi.org/10.1016/ S2213-2600(14)70290-5
11. Scherag A, Schöneweck F, Kesselmeier M, Taudien S, Platzer M, Felder M, Sponholz C, Rautanen A, Hill AVS, Hinds CJ, Hossain H, Suttorp N, Kurzai O, Slevogt H, Giamarellos-Bourboulis EJ, Armaganidis A, Trips E, Scholz M, Brunkhorst FM (2016) Genetic factors of the disease course after sepsis: a genome-wide study for 28 day mortality. EBioMedicine 12:239–246. https://doi.org/10.1016/j.ebiom.2016.08.043
12. Lu H, Wen D, Wang X, Gan L, Du J, Sun J, Zeng L, Jiang J, Zhang A (2019) Host genetic variants in sepsis risk: A field synopsis and meta-analysis. Crit Care. https://doi.org/10.1186/s13054-019-2313-0
13. Jones PA (2012) Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nat Rev Genet 13:484–492. https://doi.org/10.1038/nrg3230
14. Binnie A, Walsh CJ, Hu P, Dwivedi DJ, Fox-Robichaud A, Liaw PC, Tsang JLY, Batt J, Carrasqueiro G, Gupta S, Marshall JC, Castelo-Branco P, Dos Santos CC (2020) Epigenetic profiling in severe sepsis: a pilot study of DNA methylation profiles in critical illness. Crit Care Med 48:142–150. https://doi.org/10.1097/CCM.0000000000004097
15. Lorente-Sorolla C, Garcia-Gomez A, Català-Moll F, Toledano V, Ciudad L, Avendaño-Ortiz J, Maroun-Eid C, MartínQuirós A, Martínez-Gallo M, Ruiz-Sanmartín A, Del Campo ÁG, Ferrer-Roca R, Ruiz-Rodriguez JC, Álvarez-Errico D, López-Collazo E, Ballestar E (2019) Inflammatory cytokines and organ dysfunction associate with the aberrant DNA methylome of monocytes in sepsis. Genome Med 11:66. https://doi.org/10.1186/s13073-019-0674-2
16. Scicluna BP, Uhel F, van Vught LA, Wiewel MA, Hoogendijk AJ, Baessman I, Franitza M, Nürnberg P, Horn J, Cremer OL, Bonten MJ, Schultz MJ, van der Poll T, consortium MD and RS in S (MARS), (2020) The leukocyte non-coding RNA landscape in critically ill patients with sepsis. Elife 9:e58597. https://doi.org/10.7554/eLife.58597
17. Stark R, Grzelak M, Hadfield J (2019) RNA sequencing: the teenage years. Nat Rev Genet 20:631–656. https://doi.org/10.1038/s41576-019-0150-2
18. Stanski NL, Wong HR (2020) Prognostic and predictive enrichment in sepsis. Nat Rev Nephrol 16:20–31. https://doi.org/10.1038/s41581-019-0199-3
19. Sweeney TE, Wong HR, Khatri P (2016) Robust classification of bacterial and viral infections via integrated host gene expression diagnostics. Sci Transl Med. https://doi.org/10.1126/scitranslmed.aaf7165
20. McHugh L, Seldon TA, Brandon RA, Kirk JT, Rapisarda A, Sutherland AJ, Presneill JJ, Venter DJ, Lipman J, Thomas MR, Klein Klouwenberg PMC, van Vught L, Scicluna B, Bonten M, Cremer OL, Schultz MJ, van der Poll T, Yager TD, Brandon RB (2015) A molecular host response assay to discriminate between sepsis and infection-negative systemic inflammation in critically ill patients: discovery and validation in independent cohorts. PLoS Med. https://doi.org/10.1371/ journal.pmed.1001916
21. Scicluna BP, Klein Klouwenberg PMC, Van Vught LA, Wiewel MA, Ong DSY, Zwinderman AH, Franitza M, Toliat MR, Nürnberg P, Hoogendijk AJ, Horn J, Cremer OL, Schultz MJ, Bonten MJ, Van Der Poll T (2015) A molecular biomarker to diagnose community-acquired pneumonia on intensive care unit admission. Am J Respir Crit Care Med 192:826–835. https://doi.org/10.1164/rccm.201502-0355OC
22. Wong HR, Cvijanovich NZ, Anas N, Allen GL, Thomas NJ, Bigham MT, Weiss SL, Fitzgerald J, Checchia PA, Meyer K, Shanley TP, Quasney M, Hall M, Gedeit R, Freishtat RJ, Nowak J, Shekhar RS, Gertz S, Dawson E, Howard K, Harmon K, Beckman E, Frank E, Lindsell CJ (2015) Developing a clinically feasible personalized medicine approach to pediatric septic shock. Am J Respir Crit Care Med 191:309–315. https://doi.org/10.1164/rccm.201410-1864OC
23. Davenport EE, Burnham KL, Radhakrishnan J, Humburg P, Hutton P, Mills TC, Rautanen A, Gordon AC, Garrard C, Hill AVS, Hinds CJ, Knight JC (2016) Genomic landscape of the individual host response and outcomes in sepsis: A prospective cohort study. Lancet Respir Med 4:259–271. https://doi.org/10.1016/S2213-2600(16)00046-1
24. Scicluna BP, van Vught LA, Zwinderman AH, Wiewel MA, Davenport EE, Burnham KL, Nürnberg P, Schultz MJ, Horn J, Cremer OL, Bonten MJ, Hinds CJ, Wong HR, Knight JC, van der Poll T, on behalf of the MARS consortium (2017) Classification of patients with sepsis according to blood genomic endotype: a prospective cohort study. Lancet Respir Med 5:816–826. https://doi.org/10.1016/S2213-2600(17)30294-1
25. Sweeney TE, Azad TD, Donato M, Haynes WA, Perumal TM, Henao R, Bermejo-Martin JF, Almansa R, Tamayo E, Howrylak JA, Choi A, Parnell GP, Tang B, Nichols M, Woods CW, Ginsburg GS, Kingsmore SF, Omberg L, Mangravite LM, Wong HR, Tsalik EL, Langley RJ, Khatri P (2018) Unsupervised analysis of transcriptomics in bacterial sepsis across multiple datasets reveals three robust clusters. Crit Care Med 46:915–925. https://doi.org/10.1097/ccm.0000000000 003084
26. DeMerle KM, Angus DC, Baillie JK, Brant E, Calfee CS, Carcillo J, Chang C-CH, Dickson R, Evans I, Gordon AC, Kennedy J, Knight JC, Lindsell CJ, Liu V, Marshall JC, Randolph AG, Scicluna BP, Shankar-Hari M, Shapiro NI, Sweeney TE, Talisa VB, Tang B, Thompson BT, Tsalik EL, van der Poll T, van Vught LA, Wong HR, Yende S, Zhao H, Seymour CW (2021) Sepsis subclasses: a framework for development and interpretation. Crit Care Med. https://doi.org/10.1097/CCM. 0000000000004842
27. Antcliffe DB, Burnham KL, Al-Beidh F, Santhakumaran S, Brett SJ, Hinds CJ, Ashby D, Knight JC, Gordon AC (2019) Transcriptomic signatures in sepsis and a differential response to steroids from the VaNISH randomized trial. Am J Respir Crit Care Med 199:980–986. https://doi.org/10.1164/rccm.201807-1419OC
28. Wong HR, Atkinson SJ, Cvijanovich NZ, Anas N, Allen GL, Thomas NJ, Bigham MT, Weiss SL, Fitzgerald JC, Checchia PA, Meyer K, Quasney M, Hall M, Gedeit R, Freishtat RJ, Nowak J, Raj SS, Gertz S, Lindsell CJ (2016) Combining prognostic and predictive enrichment strategies to identify children with septic shock responsive to corticosteroids. Crit Care Med 44:e1000–e1003. https://doi.org/10.1097/CCM.0000000000001833
29. Claushuis TAM, Van Vught LA, Scicluna BP, Wiewel MA, Klein Klouwenberg PMC, Hoogendijk AJ, Ong DSY, Cremer OL, Horn J, Franitza M, Toliat MR, Nürnberg P, Zwinderman AH, Bonten MJ, Schultz MJ, Van Der Poll T (2016) Thrombocytopenia is associated with a dysregulated host response in critically ill sepsis patients. Blood 127:3062–3072. https:// doi.org/10.1182/blood-2015-11-680744
30. Zador Z, Landry A, Balas M, Marshall JC, Cusimano MD (2020) Data driven analysis reveals shared transcriptome response, immune cell composition, and distinct mortality rates across differing etiologies of critical illness. Crit Care Med 48:338–343. https://doi.org/10.1097/CCM.0000000000004128
31. Newman AM, Liu CL, Green MR, Gentles AJ, Feng W, Xu Y, Hoang CD, Diehn M, Alizadeh AA (2015) Robust enumeration of cell subsets from tissue expression profiles. Nat Methods 12:453–457. https://doi.org/10.1038/nmeth.3337
32. Salomon R, Kaczorowski D, Valdes-Mora F, Nordon RE, Neild A, Farbehi N, Bartonicek N, Gallego-Ortega D (2019) Droplet-based single cell RNAseq tools: a practical guide. Lab Chip 19:1706–1727. https://doi.org/10.1039/c8lc0 1239c
33. Reyes M, Filbin MR, Bhattacharyya RP, Billman K, Eisenhaure T, Hung DT, Levy BD, Baron RM, Blainey PC, Goldberg MB, Hacohen N (2020) An immune-cell signature of bacterial sepsis. Nat Med 26:333–340. https://doi.org/10.1038/ s41591-020-0752-4
34. Wen M, Cai G, Ye J, Liu X, Ding H, Zeng H (2020) Single-cell transcriptomics reveals the alteration of peripheral blood mononuclear cells driven by sepsis. Ann Transl Med 8:125–125. https://doi.org/10.21037/atm.2020.02.35
35. Jiang Y, Rosborough BR, Chen J, Das S, Kitsios GD, McVerry BJ, Mallampalli RK, Lee JS, Ray A, Chen W, Ray P (2020) Single cell RNA sequencing identifies an early monocyte gene signature in acute respiratory distress syndrome. JCI Insight. https://doi.org/10.1172/jci.insight.135678
36. Cao Z, Robinson RAS (2014) The role of proteomics in understanding biological mechanisms of sepsis. Proteomics Clin Appl 8:35–52. https://doi.org/10.1002/prca.201300101
37. Pierrakos C, Velissaris D, Bisdorff M, Marshall JC, Vincent JL (2020) Biomarkers of sepsis: Time for a reappraisal. Crit Care 24:1–15. https://doi.org/10.1186/s13054-020-02993-5
38. van Engelen TSR, Wiersinga WJ, Scicluna BP, van der Poll T (2018) Biomarkers in Sepsis. Crit Care Clin 34:139–152. https://doi.org/10.1016/j.ccc.2017.08.010
39. De Coux A, Tian Y, Deleon-Pennell KY, Nguyen NT, De Castro Brás LE, Flynn ER, Cannon PL, Griswold ME, Jin YF, Puskarich MA, Jones AE, Lindsey ML (2015) Plasma glycoproteomics reveals sepsis outcomes linked to distinct proteins in common pathways. Crit Care Med 43:2049–2058. https://doi.org/10.1097/CCM.0000000000001134
40. Cao Z, Yende S, Kellum JA, Angus DC, Robinson RAS (2014) Proteomics reveals age-related differences in the host immune response to sepsis. J Proteome Res 13:422–432. https://doi.org/10.1021/pr400814s
41. Langley RJ, Tsalik EL, Van Velkinburgh JC, Glickman SW, Rice BJ, Wang C, Chen B, Carin L et al (2013) Sepsis: An integrated clinico-metabolomic model improves prediction of death in sepsis. Sci Transl Med 5:195ra95. https://doi.org/10.1126/scitranslmed.3005893
42. Sharma NK, Tashima AK, Brunialti MKC, Ferreira ER, Torquato RJS, Mortara RA, MacHado FR, Assuncao M, Rigato O, Salomao R (2017) Proteomic study revealed cellular assembly and lipid metabolism dysregulation in sepsis secondary to community-acquired pneumonia. Sci Rep 7:15606. https://doi.org/10.1038/s41598-017-15755-1
43. Su L, Cao L, Zhou R, Jiang Z, Xiao K, Kong W, Wang H, Deng J, Wen B, Tan F, Zhang Y, Xie L (2013) Identification of novel biomarkers for sepsis prognosis via urinary proteomic analysis using iTRAQ Labeling and 2D-LC-MS/MS. PLoS ONE. https://doi.org/10.1371/journal.pone.0054237
44. Liu J, Li J, Deng X (2014) Proteomic analysis of differential protein expression in platelets of septic patients. Mol Biol Rep 41:3179–3185. https://doi.org/10.1007/s11033-014-3177-7
45. Malmström E, Davidova A, Mörgelin M, Linder A, Larsen M, Qvortrup K, Nordenfelt P, Shannon O, Dzupova O, Holub M, Malmström J, Herwald H (2014) Targeted mass spectrometry analysis of neutrophil-derived proteins released during sepsis progression. Thromb Haemost 112:1230–1243. https://doi.org/10.1160/TH14-04-0312
46. Zhang C, Wang K, Yang L, Liu R, Chu Y, Qin X, Yang P, Yu H (2018) Lipid metabolism in inflammation-related diseases. Analyst 143:4526–4536. https://doi.org/10.1039/c8an01046c
47. Han X (2016) Lipidomics for studying metabolism. Nat Rev Endocrinol 12:668–679. https://doi.org/10.1038/nrendo.2016.98
48. Trinder M, Genga KR, Kong HJJ, Blauw LL, Lo C, Li X, Cirstea M, Wang Y, Rensen PCN, Russell JA, Walley KR, Boyd JH, Brunham LR (2019) Cholesteryl ester transfer protein influences high-density lipoprotein levels and survival in sepsis. Am J Respir Crit Care Med 199:854–862. https://doi.org/10.1164/rccm.201806-1157OC
49. Jones TK, Wong HR, Meyer NJ (2019) HDL cholesterol: A “pathogen lipid sink” for sepsis? Am J Respir Crit Care Med 199:812–814. https://doi.org/10.1164/rccm.201811-2084ED
50. Mecatti GC, Sánchez-Vinces S, Fernandes AMAP, Messias MCF, de Santis GKD, Porcari AM, Marson FAL, de Carvalho P, O, (2020) Potential lipid signatures for diagnosis and prognosis of sepsis and systemic inflammatory response syndrome. Metabolites 10:1–17. https://doi.org/10.3390/metabo10090359
51. Cambiaghi A, Pinto BB, Brunelli L, Falcetta F, Aletti F, Bendjelid K, Pastorelli R, Ferrario M (2017) Characterization of a metabolomic profile associated with responsiveness to therapy in the acute phase of septic shock. Sci Rep. https:// doi.org/10.1038/s41598-017-09619-x
52. Dennis EA, Norris PC (2015) Eicosanoid storm in infection and inflammation. Nat Rev Immunol 15:511–523. https:// doi.org/10.1038/nri3859
53. Chiang N, Fredman G, Bäckhed F, Oh SF, Vickery T, Schmidt BA, Serhan CN (2012) Infection regulates pro-resolving mediators that lower antibiotic requirements. Nature 484:524–528. https://doi.org/10.1038/nature11042
54. Dalli J, Colas RA, Quintana C, Barragan-Bradford D, Hurwitz S, Levy BD, Choi AM, Serhan CN, Baron RM (2017) Human sepsis eicosanoid and proresolving lipid mediator temporal profiles: correlations with survival and clinical outcomes. Crit Care Med 45:58–68. https://doi.org/10.1097/CCM.0000000000002014
55. O’Neill LAJ, Kishton RJ, Rathmell J (2016) A guide to immunometabolism for immunologists. Nat Rev Immunol 16:553–565. https://doi.org/10.1038/nri.2016.70
56. Van den Bossche J, O’Neill LA, Menon D (2017) Macrophage immunometabolism: where are we (going)? Trends Immunol 38:395–406. https://doi.org/10.1016/j.it.2017.03.001
57. Khaliq W, Großmann P, Neugebauer S, Kleyman A, Domizi R, Calcinaro S, Brealey D, Gräler M, Kiehntopf M, Schäuble S, Singer M, Panagiotou G, Bauer M (2020) Lipid metabolic signatures deviate in sepsis survivors compared to nonsurvivors. Comput Struct Biotechnol J 18:3678–3691. https://doi.org/10.1016/j.csbj.2020.11.009
58. van der Meer-Janssen YPM, van Galen J, Batenburg JJ, Helms JB (2010) Lipids in host-pathogen interactions: pathogens exploit the complexity of the host cell lipidome. Prog Lipid Res 49:1–26. https://doi.org/10.1016/j.plipres.2009.07.003
59. Chatterjee S, Kawahara R, Tjondro HC, Shaw DR, Nenke MA, Torpy DJ, Thaysen-Andersen M (2021) Serum N-Glycomics Stratifies Bacteremic Patients Infected with Different Pathogens. J. Clin, Med, p 10
60. Sender R, Fuchs S, Milo R (2016) Are we really vastly outnumbered? Revisiting the ratio of bacterial to host cells in humans. Cell 164:337–340. https://doi.org/10.1016/j.cell.2016.01.013
61. Haak BW, Wiersinga WJ (2017) The role of the gut microbiota in sepsis. Lancet Gastroenterol Hepatol 2:135–143. https://doi.org/10.1016/S2468-1253(16)30119-4
62. Prescott HC, Dickson RP, Rogers MAM, Langa KM, Iwashyna TJ (2015) Hospitalization type and subsequent severe sepsis. Am J Respir Crit Care Med 192:581–588. https://doi.org/10.1164/rccm.201503-0483OC
63. Lankelma JM, van Vught LA, Belzer C, Schultz MJ, van der Poll T, de Vos WM, Wiersinga WJ (2017) Critically ill patients demonstrate large interpersonal variation in intestinal microbiota dysregulation: a pilot study. Intensive Care Med 43:59–68. https://doi.org/10.1007/s00134-016-4613-z
64. Liu Z, Li N, Fang H, Chen X, Guo Y, Gong S, Niu M, Zhou H, Jiang Y, Chang P, Chen P (2019) Enteric dysbiosis is associated with sepsis in patients. FASEB J 33:12299–12310. https://doi.org/10.1096/fj.201900398RR
65. Agudelo-Ochoa GM, Valdés-Duque BE, Giraldo-Giraldo NA, Jaillier-Ramírez AM, Giraldo-Villa A, Acevedo-Castaño I, Yepes-Molina MA, Barbosa-Barbosa J, Benítez-Paéz A (2020) Gut microbiota profiles in critically ill patients, potential biomarkers and risk variables for sepsis. Gut Microbes 12:1707610. https://doi.org/10.1080/19490976.2019.1707610
66. McDonald D, Ackermann G, Khailova L, Baird C, Heyland D, Kozar R, Lemieux M, Derenski K, King J, Vis-Kampen C, Knight R, Wischmeyer PE (2016) Extreme dysbiosis of the microbiome in critical illness. Sphere. https://doi.org/10. 1128/msphere.00199-16
67. Dickson RP, Erb-Downward JR, Martinez FJ, Huffnagle GB (2016) The microbiome and the respiratory tract. Annu Rev Physiol 78:481–504. https://doi.org/10.1146/annurev-physiol-021115-105238
68. Dickson RP, Singer BH, Newstead MW, Falkowski NR, Erb-Downward JR, Standiford TJ, Huffnagle GB (2016) Enrichment of the lung microbiome with gut bacteria in sepsis and the acute respiratory distress syndrome. Nat Microbiol 1:16113. https://doi.org/10.1038/nmicrobiol.2016.113
69. Sepsis Lung Microbiome Study Group (2020) Could lung bacterial dysbiosis predict ICU mortality in patients with extra-pulmonary sepsis? A proof-of-concept study. Intensive Care Med 46:2118–2120. https://doi.org/10.1007/ s00134-020-06190-4
70. Greer R, Dong X, Morgun A, Shulzhenko N (2016) Investigating a holobiont: Microbiota perturbations and transkingdom networks. Gut Microbes 7:1–10. https://doi.org/10.1080/19490976.2015.1128625
71. Quince C, Walker AW, Simpson JT, Loman NJ, Segata N (2017) Shotgun metagenomics, from sampling to analysis. Nat Biotechnol 35:833–844. https://doi.org/10.1038/nbt.3935
72. Moss EL, Maghini DG, Bhatt AS (2020) Complete, closed bacterial genomes from microbiomes using nanopore sequencing. Nat Biotechnol 38:701–707. https://doi.org/10.1038/s41587-020-0422-6
73. Haak BW, Prescott HC, Wiersinga WJ (2018) Therapeutic potential of the gut microbiota in the prevention and treatment of sepsis. Front Immunol 9:2042. https://doi.org/10.3389/fimmu.2018.02042
74. Huang S, Chaudhary K, Garmire LX (2017) More is better: Recent progress in multi-omics data integration methods. Front Genet 8:84. https://doi.org/10.3389/fgene.2017.00084
75. Subramanian I, Verma S, Kumar S, Jere A, Anamika K (2020) Multi-omics data integration, interpretation, and its application. Bioinform Biol Insights 14:1177932219899051. https://doi.org/10.1177/1177932219899051
76. Wong HR, Cvijanovich NZ, Anas N, Allen GL, Thomas NJ, Bigham MT, Weiss SL, Fitzgerald JC, Checchia PA, Meyer K, Quasney M, Hall M, Gedeit R, Freishtat RJ, Nowak J, Raj SS, Gertz S, Grunwell JR, Lindsell CJ (2017) Improved risk stratification in pediatric septic shock using both protein and mRNA Biomarkers: Persevere-XP. Am J Respir Crit Care Med 196:494–501. https://doi.org/10.1164/rccm.201701-0066OC
77. Wang L, Ko ER, Gilchrist JJ, Pittman KJ, Rautanen A, Pirinen M, Thompson JW, Dubois LG, Langley RJ, Jaslow SL, Salinas RE, Rouse DC, Moseley MA, Mwarumba S, Njuguna P, Mturi N, Williams TN, Scott JAG, Hill AVS, Woods CW, Ginsburg GS, Tsalik EL, Ko DC (2017) Human genetic and metabolite variation reveals that methylthioadenosine is a prognostic biomarker and an inflammatory regulator in sepsis. Sci Adv 3:e1602096. https://doi.org/10.1126/sciadv. 1602096
Schuurman et al. Intensive Care Medicine Experimental (2021) 9:27