Новые микробиологические технологии для диагностики бактериальной инфекции и сепсиса
Current Infectious Disease Reports (2021) 23:12
Новые микробиологические технологии для диагностики бактериальной инфекции и сепсиса в отделении интенсивной терапии, включая технологии «Point-of-Care»
Перевод оригинальной статьи «New Microbiological Techniques for the Diagnosis of Bacterial Infections and Sepsis in ICU Including Point-of-Care»
Авторы: Anna Maria Peri, Adam Stewart, Anna Hume, Adam Irwin, Patrick N. A. Harris
University of Queensland Centre for Clinical Research, Herston, Queensland 4029, Australia
Royal Brisbane and Women’s Hospital, Queensland Health, Herston, Queensland, Australia Central Microbiology, Pathology Queensland, Royal Brisbane and Women’s Hospital, Herston, Queensland 4029, Australia
Queensland Children’s Hospital, QLD, Brisbane, Australia
Введение
Бактериальные инфекции распространены у взрослых и детей, госпитализированных в отделения интенсивной терапии (ОИТ). В когорте из 198 ОИТ из 24 стран Европы, в которую вошли 3 147 пациентов, у 37.4% был установлен диагноз сепсис, а у 24.7% диагноз сепсиса устанавливался на момент госпитализации в ОИТ [1]. Инфекции у таких пациентов ассоциировались со значительным ростом заболеваемости и летальности, со значительным ростом стоимости лечения [2]. К рискам, что связаны с развитием инфекции, относят и широкое применение антибиотиков. К примеру, в глобальном исследовании распространенности было установлено, что 70% от всех пациентов в ОИТ получали хотя бы один антибиотик в любой день исследования [3]. Возможность быстро и точно определить возбудителя (патогена) бактериальной инфекции является чрезвычайно важным условием эффективности клинического менеджмента. Более того, в условиях растущего бремени, что накладывает на системы здравоохранения антимикробная резистентность, быстрый тест на антимикробную чувствительность (англ. antimicrobial susceptibility testing) приобретает критическую важность для управления антимикробной терапией. Учитывая необходимость в снижении чрезмерного применения антибиотиков, мы также нуждаемся в стратегиях диагностики, что смогут помочь исключить наличие инфекции, тем самым определить состояние воспаления не инфекционной природы, при котором применение антибиотиков не требуется [4].
Существующие на сегодня диагностические методы у пациентов с сепсисом больше полагаются на посев культуры микроорганизмов из пробы крови для определения бактериемии. Но всем нам известно, что такой подход не только относительно длительный в контексте получения результата, он еще и трудоемкий, а сами системы, основанные на посеве культуры, страдают от ряда пре-аналитических ограничений, которые могут отрицательно повлиять на результат, например, недостаточный объем крови для посева, предшествующее анализу воздействие антибиотиков и задержки при транспортировке проб в лабораторию, особенно в случаях, когда лаборатория находится вдали от места взятия проб на культуральные посевы. К тому же, даже если был получен положительный результат в виде роста некоего микроорганизма, точная его идентификация и тест на чувствительность к антибиотикам может занять несколько дней. Также довольно частая проблема - контаминация проб крови, что лежит в основе, или даже является той силой, что приводит к ненадлежащему назначению антибиотиков, неправильной диагностике и увеличивает время воздействия токсичных эффектов, связанных с антибиотиками [5]. Также не станем забывать о «привередливых» патогенах, обеспечение роста которых в стандартных автоматических системах вызывает затруднения [6].
В некотором отношении микробиологические лаборатории в течение длительного времени полагались на технологии, которые практически не менялись, и это в то время, когда все больше появляется новейших технологий, что способны совсем уже в ближайшем будущем произвести революцию в микробной диагностике. Еще десять лет назад методы масс-спектрометрии не были частью рутинной лабораторной практики, а сейчас в практику пришла масс-спектрометрия с лазерной десорбцией/ионизацией с использованием матрицы (англ. matrix-assisted laser desorption/ionisation time-of-flight mass spectrometry или MALDI-TOF), которая во многих лабораториях очень быстро заменила обычные методы идентификации бактерий, но уже со уменьшением длительности анализа при увеличении его точности и все это происходит с одновременным снижением затрат [7]. Эта статья направлена на обзор современного состояния микробной диагностики, а также новых технологий, которые смогут улучшить наши возможности в быстрой и точной микробиологической диагностики у пациентов с бактериальными инфекциями и сепсисом.
Установленные методы быстрой диагностики
За последние несколько лет внедрение быстрых диагностических технологий в лабораторную практику и улучшения в автоматизации рабочего процесса автоматических систем позволило улучшить качество оказания медицинской помощи [8, 9]. В настоящее время автоматические системы посева крови стали золотым стандартом при выявлении инфекций кровотока [10]. Достаточно много систем для автоматизированного роста культуры крови (типа BACTEC TM FX; BacT/ALERT®), в которых применяются различные методы для определения роста микроорганизмов (например, разные нутриенты и антимикробные связывающие агенты) [11—15], а производительность этих систем уже была подвергнута сравнению. Флаконы, которые содержат связывающие антибиотики агенты, обычно показывают лучшую скорость роста микроорганизмов [16, 17]. Большая часть автоматических систем для роста культуры крови имеют в своем составе внутренний датчик, который определяет углекислый газ или рН, как индикаторы роста микробов [18]. Микроскопия и окраска по Граму в стерильных жидкостях, какой, например является кровь, важный шаг в предоставлении информации для формирования правильного диагноза и последующего надлежащего менеджмента тяжелой инфекции. Несмотря на автоматизацию окраски по Граму, интерпретация результатов все еще остается трудоемким процессом, занимающем много времени, и все еще этот процесс зависит от оператора [19]. И хотя уже были изучены подходы к автоматизированной классификации окраски по Граму, основанные на автоматическом получении изображений и машинном обучении и показавшие многообещающую точность, они очень далеки от возможности работать в виде полностью автоматизированных систем. Ранняя идентификация таких микроорганизмов как Staphylococcus aureus напрямую с использованием коагулазного теста показало снижение времени на получение результата и оказалось относительно недорогим методом, который может быть внедрен в стандартные рабочие процессы [21, 22]. Быстрые методы определения антимикробной чувствительности также привели к снижению времени получения результата. Европейский комитет по тестам антимикробной чувствительности (The European Committee on Antimicrobial Susceptibility Testing или EUCAST) разработал стандартизированный быстрый метод, основанный на дисковой диффузии, который дает результаты антимикробной чувствительности в течение 2—8 часов после начала роста культуры крови [23—25]. Также возможно сегодня и определение основных медиаторов резистентности, таких как β-лактамазы, напрямую в клинической пробе. Быстрое определение клинически значимых микроорганизмов, продуцирующих карбапенемазы и β-лактамазы расширенного спектра (ESBL), может быть сделано с помощью множества коммерчески доступных тестов (например, RAPIDEC® CARBA NP, ESBL-NP) [26, 27]. Использование рН индикатора для определения гидролиза карбапенема привело к быстрому (менее двух часов) и точному определению карбапенемаз в клинических изолятах при достаточно небольшом времени инкубации культуры [28]. Аналогичное снижение времени на получение результатов было показано и при использовании тестов на определение ESBL при сравнении их со стандартными методами тестирования на чувствительность [29].
Прямой тест клинических проб на антиген нацелен на быструю идентификацию вида микроорганизма. Тестирование мочи на антиген широко используется для определения инфекций респираторного тракта, вызванных Legionella pneumophila и Streptococcus pneumoniae [30]. Антигены, выделяемые этими микроорганизмами в мочевыводящие пути, обычно обнаруживаются с помощью иммуноферментного анализа (enzyme immunoassay или EIA) или анализа латерального потока (lateral flow assay или LFA) [31]. Но несмотря на укороченное время получения результатов, тестирование на антигены обладает слабой чувствительностью и специфичностью (например, ребенок с колонизацией S. pneumoniae), что делает невозможным их применение для определения профиля чувствительности к антибиотикам и для других эпидемиологических целей [32]. Быстрая детекция антигена в других клинических пробах, как кровь, мазок ротоглотки, синовиальная жидкость, плевральный выпот, цереброспинальная жидкость ранее была изучена, но широкого применения в рутинной клинической практике не получила. Большое ретроспективное исследование, в котором оценивалась клиническая полезность быстрой детекции бактериального антигена с использованием латексной агглютинации, пришло к выводу, что данный метод требует больших затрат без какой-либо клинической пользы [36]. Тестирование амплификации нуклеиновых кислот (англ. Nucleic acid amplification testing или NAAT) или полимеразная цепная реакция (PCR) полезны при методах детекции микробов без предварительного посева и часто используются в лабораториях всего мира для диагностики широкого спектра микробных патогенов. К тому же, при мультиплексной PCR в одной реакционной пробирке содержатся несколько праймеров и датчиков (зондов) для амплификации генов-мишеней от множества патогенов [37]. Такой высоко чувствительный подход увеличивает диагностическую ценность метода и может использоваться для многих клинических образцов, включая сюда секрет дыхательных путей, цереброспинальную жидкость, стерильные жидкости и каловые массы при диарее. К ограничениям PCR относят сообщения о побочных/непрофильных результатах, невозможность отделить колонизацию от инфекции, требование к опыту оператора, требование к лабораторной среде и отсутствие данных о чувствительности к антибиотикам. Более того, при PCR определяются только те патогены, на которые настроена цель анализа. Редко встречающиеся микроорганизмы, а также те, на которые тест не настроен (неожиданные) или те, штаммы которых изменили последовательности, могут быть пропущены.
Иммунный ответ на тяжелую инфекцию и сепсис является комплексным и приводит к продукции разнообразных воспалительных и противовоспалительных медиаторов [39]. Часть из биомаркеров уже изучены в контексте быстрой диагностики тяжелой инфекции в ОИТ. Наряду с количеством лейкоцитов, наиболее известным из них является С-реактивный белок (СРБ). СРБ относится к белкам острой фазы, уровень которого увеличивается вслед за секрецией интерлейкина (IL) 6 макрофагами и Т-клетками, и показал себя чувствительным, но не специфичным, маркером сепсиса [40]. Прокальцитонин - пептид, секреция которого осуществляется множеством клеток в ответ на провоспалительные стимулы, может быть более специфичным маркером бактериальной инфекции, чем СРБ [41]. Комплексность и сложность иммунного ответа отражается диапазоном биомаркеров, которые исследуются как потенциальные биомаркеры тяжелой инфекции, и в этот диапазон входят белки острой фазы, цитокины (в основном IL-6 и IL-8), водорастворимые рецепторы и маркеры целостности клеток и эндотелия [42]. Используя их поодиночке или в сочетаниях, роль биомаркеров сводится к стратификации риска развития тяжелой инфекции или к грубому прогнозированию вероятной этиологии, а также к помощи в выборе начальной терапии антибиотиками или к ее прекращению. Несмотря на ограничения при применении биомаркеров для прогнозирования, данные рандомизированных исследований подтверждают их значение для применения в условиях ОИТ. В то время, как они имеют ограниченную ценность для решения вопроса о начале антибиотикотерапии, применение прокальцитонина для решения вопроса о возможном прекращении терапии антибиотиками снизило продолжительность ее в ОИТ как у взрослых, так и у новорожденных [43, 44].
Какие методы на подходе к клинической практике
За последние годы стали появляться новые тесты быстрой диагностики, при которых становится возможным не только идентификация патогена, но и выяснение профиля его резистентности за сравнительно непродолжительный период. Все это вселяет в нас уверенность в скором улучшении менеджмента пациента, даже несмотря на то, что данные по их клиническому влиянию еще недостаточны [45].
Новые методы диагностики менингита и респираторных инфекций
Растет частота применения мультиплексной PCR в отделениях интенсивной терапии для диагностики инфекций центральной нервной системы и пневмонии. К примеру, панель «bioMerieux» (the BioFire FilmArray Meningitis/ Encephalitis panel) уже получила одобрение FDA, способна определить 14 патогенов ЦНС за один час. Установленная чувствительность и специфичность составляет 90% и 97% соответственно [46], но доказательства влияния их на клинические исходы все еще скудные [47, 48]. Подобным образом панель «the BioFire FilmArray Pneumonia plus» способна определить 27 микроорганизмов и 7 маркеров резистентности из проб, взятых в дыхательных путях, включая сюда и нозокомиальные патогены, ассоциированные с внутрибольничной пневмонией и вентилятор-ассоциированной пневмонией. Уже показана роль такого подхода в улучшении контроля за назначением антибиотиков у пациентов, находящихся в критическом состоянии, с новой коронавирусной инфекцией 2019 [49]. У других мультиплексных PCR, к примеру панель Seegene Allplex Respiratory panel, более узкие задачи - здесь панель предназначена только для детекции внебольничных респираторных инфекций [50].
Детекция нуклеиновых кислот из культуры крови
Недавно появилось несколько технологий для идентификации патогена из положительного посева крови. Идентификационная панель «The BioFire FilmArray BC identification panel» (BCID, bioMérieux) основана на технологии мультиплексной PCR и способна определять 24 патогена и 3 гена резистентности в положительном посеве крови с хорошей аналитической производительностью [51, 51]. В исследовании пациентов ОИТ с сепсисом, подтвержденным положительными посевами, этот тест снижал время до начала оптимального лечения по сравнению со стандартными посевом крови с подтверждением высокой роли данного теста в диагностике вентилятор-ассоциированной пневмонии [54]. Недавно была выпущено новая версия этого теста - BioFire BCID2, с более широкой панелью, которая включает в себя 43 цели, но этот тест все еще ждет подтверждения своей полезности в клинической практике.
Касаемо такой специфической инфекции, как бактериемия S. aureus - с помощью анализатора PCR «the Xpert MRSA/SA BC Assay» (Cepheid) в реальном времени можно определить какой золотистый стафилококк, метициллин чувствительный или метициллин-резистентный (MRSA), ответственен за положительный посев крови [55], а полностью автоматизированный процесс очень легко вписывается в рутинную лабораторную практику. Подобным образом работает и анализатор «Xpert Carba-R» (Cepheid), предназначенный для определения генов, отвечающих за продукцию карбапенемаз в бактериальном изоляте, но данный метод больше подходит для ситуаций, когда превалирует высокая резистентность к карбапенемам [56]. Система «the Verigene» (Luminex) использует мультиплексную PCR и последующую гибридизацию микрочипов для определения 22 бактерий и их детерминант резистентности из положительного посева крови [57, 58] и включает в себя две панели, одна для Грамположительных микроорганизмов, а другая для Грамотрицательных. Выбор между Грам+ и Грам- определяется результатами окрашивания. Система «Verigene» уже доказала свою способность к идентификации чувствительности к новым β - лактам/β - лактамаз ингибиторам [59], что при внедрении этого метода в программы контроля за назначением антибиотиков привело к снижению времени до назначения оптимальной терапии у пациентов с бактериемией [60, 61].
К другим технологиям, полагающимся на положительные посевы крови, можно отнести флюоресцентную гибридизацию in situ (FISH), при которой используется датчик нуклеиновой кислоты (англ. nucleic acid probes или PNA), нацеленный на 16S или 18S rRNA бактерий или грибов соответственно. PNA-FISH (AdvanDx) содержит четыре разные панели и также приводит к улучшению в виде раннего надлежащего назначения антибиотиков [62, 63].
Прямая детекция патогена в крови
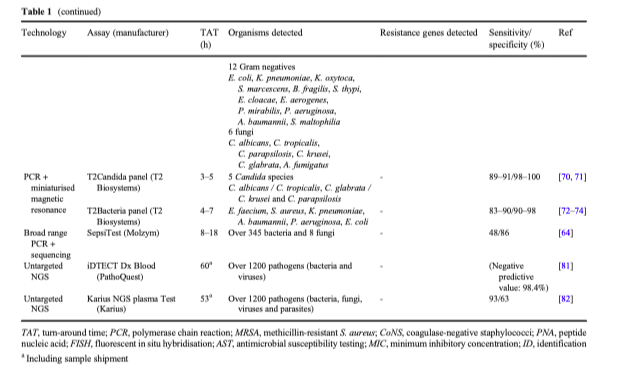
Для того, чтобы иметь возможность пропустить такой требующий времени шаг, как ожидание роста гемокультуры, появились новые технологии, способные определить патоген в пробе цельной крови. Среди них тесты Lightcycler SeptiFast (Roche) и Magicplex Sepsis Real-Time (Seegene), основанные на PCR в реальном времени, при которой определяются несколько микроорганизмов и некоторые маркеры резистентности в цельной крови. Несмотря на широкий выбор панелей, сохраняющаяся низкая чувствительность делает практически невозможным рекомендации для применения их в клинической практике [64—68]. Комбинация специфичной для патогенов PCR с миниатюрным магнитным резонансом была реализована в магнитном резонансе Т2 (T2MR), позволяющем идентифицировать микроорганизмы в цельной крови с помощью совершенно новой методологии - ДНК, амплифицированная с помощью PCR, связывается дополнительными зондами с парамагнитными наночастицами, сигнал которых идентифицируется с помощью T2MR [69]. Тест T2Candida (T2 Biosystems) - автоматизированная система, которая идентифицирует наиболее распространённые виды Candida с высокими отрицательными прогностическими значениями в широком диапазоне предтестовых вероятностей [70, 71]. Тест T2Candida показал резкое снижение времени для назначения эффективной противогрибковой терапии и снизил частоту назначений эмпирической терапии [71]. Еще один тест, the T2Bacteria, определяет бактерии ESKAPE (Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa, Escherichia coli) [72–74], но из-за ограниченного выбора патогенов и генов резистентности, клиническая польза от его применения все еще не определена [75]. MALDI-TOF использовался непосредственно на клинических образцах, включая кровь, мочу [76] и спинномозговую жидкость [77]. ПЦР/ESI-MS сочетает в себе ПЦР, специфичную для патогенов, с масс-спектрометрией, основанной на ионизации электрораспылением (ESI-MS), а система IRIDICA (Abbott) могла обнаруживать 780 микроорганизмов и 4 гена устойчивости из различных образцов. Однако, несмотря на многообещающие характеристики, этот анализ был прекращен [78], что свидетельствует о том, что явные преимущества от внедрения этих дорогостоящих тестов еще не очевидны.
Прямая метагеномика
Анализаторы, основанные на метагеномике, сейчас представляют собой многообещающие инструменты в клинической микробиологии, позволяющие идентифицировать любой микроорганизм в какой-либо пробе. 16S метагеномика базируется на амплификации через универсальные праймеры бактериального гена 16S-рРНК с последующим секвенированием ампликонов, что ведет к бактериальной идентификации и таксономическому профилированию [79]. К примеру, SepsiTest (Molzym) является полуавтоматическим анализатором, принцип действия которого основан на технологиях, использующихся для детекции патогенов в крови. Но несмотря на способность данных тестов определять полимикробные инфекции и редкие микроорганизмы, их роль в предоставлении информации для принятия клинического решения ограничена из-за низкой чувствительности, что не позволяет из применять в программах контроля за назначением антибиотиков [64]. В сравнении с 16S метагеномикой, метод дробовика (англ. Shotgun) или «Шотган-метагеномика» основан на более современном методе секвенирования (англ. next-generation sequencing или NGS), при котором возможно прочитать полные бактериальные геномы путем параллельного секвенирования, предоставляющего точное таксономическое разрешение всех патогенов в образце, а также, потенциально, и маркеров антимикробной резистентности [80]. Тест iDTECT Dx Blood (PathoQuest) недавно показал свои возможности в определении большинства клинически значимых микроорганизмов, что превосходит возможности обычной микробиологии, особенно у иммуноскомпроментированных пациентов с высокой отрицательной прогностической ценностью [81]. Тест Karius NGS Plasma Test (Karius) может идентифицировать микробные ДНК, находящиеся вне клеток, более чем 1200 микроорганизмов, показывая при этом 97% чувствительность по сравнению с обычным посевом крови, у пациентов с сепсисом [82] и, возможно, может определять клинически значимые патогены в крови за несколько дней до развития клинической картины инфекции кровотока [83].
Возможность метагеномики дробовика выполнять всесторонний анализ микробного генетического материала в биологическом образце очень впечатляет. Но в то же время имеются и ограничения, что усложняют внедрение таких анализаторов в клиническую практику, потому что в некоторых исследованиях сообщается об ограниченном клиническом воздействии данных инструментов диагностики инфекции [84, 85]. Например, частая детекция контаминáнтов и колониезирующих инфекций оказывают неблагоприятное влияние на специфичность тестов, основанных на NGS, и затрудняют интерпретацию результатов при диагностике инфекций кровотока. Для преодоления такого ограничения в недавнем исследовании пациентов с септическим шоком была показана полезность шкалы количественной оценки сепсиса (англ. the sepsis indicating quantifier или SIQ) как средства дискриминации клинически значимых патогенов от других [86]. Более того, чувствительность методов, основанных на NGS, снижается в образцах с высоким фоном нуклеиновых кислот, таких как кровь, что требует элиминации ДНК человека. Также таким методам не хватает стандартизации анализа. Часто навыки биоинформатики, необходимые для анализа данных NGS, недоступны в стандартной диагностической лаборатории, что может потребовать обращения в другие организации или даже передачи в них клинических данных. Все это приводит к задержкам в получении результатов и к дополнительным расходам, включая сюда возможные проблемы с вычислительной мощностью и емкостью хранения данных, проблемам конфиденциальности данных и сложностям с аккредитацией в регулирующих органах.
Новые методы быстрой диагностики антимикробной чувствительности
Детекция генов резистентности не всегда надежно отражает актуальные паттерны чувствительности у идентифицированных патогенов. Система, одобренная FDA, Accelerate Pheno system (Accelerate Diagnostics) может определить 16 микроорганизмов из положительного посева крови и основана на технологии FISH, но эта же система способна определить фенотип антимикробной чувствительности путем морфокинетического клеточного анализа [87–89] с категориальным согласием в 96% по сравнению со стандартными методами [89, 90] (Таблица 1). Исследования показали, что данный тест приводит к более раннему назначению оптимальной терапии у пациентов с бактериемией [92, 93].
Развитие электроники, микрожидкостных и оптических технологий, технологий биосенсоров вселяет в нас надежду в быстрое внедрение тестов для быстрого определения антимикробной чувствительности в клиническую практику. Очевидна роль таких тестов при тестировании непосредственно у кровати пациента (англ. point of care testing или POCT), но все же клинических данных явно недостаточно и в настоящий момент идут исследования, направленные на получение одобрения от FDA и CE mark [94].
Иммунный ответ (ответ «хозяина) и транскриптомика
Существующие на сегодня биомаркеры не позволяют нам полностью заглянуть в такой сложный процесс, как иммунный ответ на инфекцию, из чего следует невозможность полного понимания этиологии инфекции. Основные биомаркеры, которые сейчас используются в клинической практике, предлагают только бинарную оценку тяжести инфекции или указывают на вероятность бактериальной инфекции, что позволяет им служить для нас в качестве «проводников» (или гидов) в менеджменте антибактериальной терапии. Это определяет растущий интерес к роли омических технологий - исследования протеома, метаболома, эпигенома или транскриптома, применение которых послужит получению более полной характеристики фенотипов инфекции. Подобные биологические классификаторы уже применяются в онкологии [95] и в терапии сердечно-сосудистых заболеваний [96], но для применения их отделениях интенсивной терапии критичный по времени характер инфекций создает определенные сложности.
Диагностика на основе дифференциальной экспрессии генов при острых инфекциях постоянно привлекает к себе внимание. В отношении таких классификаторов, как «Интегрированная модель о принятии решения по антибиотикам» (англ. Integrated Antibiotics Decision Model) [97] и «Шкала риска заболевания» (англ. Disease Risk Score) у детей с фебрильной температурой [98], после прохождения ими внешней проверки, позволило предположить об их возможной ценности при принятии клинических решений. «Septicyte» был первым тестом, основанным на транскриптомной диагностике инфекционных болезней, который получил одобрения FDA в 2017 году и в настоящий момент проходит проверку в различных клинических ситуациях [99]. Классификаторы на основе транскриптом обладают возможностью не только характеризовать пациентов по патогенам (бактерии, вирусы или грибы), но и по воспалительному фенотипу, тем самым предлагая новые возможности для проведения более персонализированной иммуномодуляции при сепсисе [100]. Роль транскриптомных классификаторов в диагностике инфекций в ОИТ требует понимания того, какие именно анализы необходимо будет делать, а также своевременность этих анализов и их влияние не только на эффективность лечения, но и на расходы, но все эти сведения мы получим после проведения клинических исследований.
Как следует оценивать клиническую полезность новой быстрой диагностики?
Снижение средней продолжительности времени для получения результата (англ. turn-around time или TAT) в виде идентификации патогена или его чувствительности не является указанием на полезность теста, даже несмотря на то, что время является важным компонентом [101]. Есть и другие параметры в виде чувствительности и специфичности полученного результата и доверия к нему со стороны клиницистов [102]. Полная оценка полезности технологий быстрой микробиологической диагностики не должна заканчиваться только на времени получения результата и на управлении антимикробной терапией. Необходимы контролируемые исследования, в которых бы оценивались множество ключевых клинических исходов - летальность, острое почечное повреждение, продолжительность лечения и повторные госпитализации. Было бы идеально, если бы клинические исследования сочетались бы с исследованиями эффективности затрат, в которых оценивалась не только стоимость лечения в госпитале, но и качество жизни после выписки из стационара и продолжительность жизни.
Мы испытываем крайнюю скудность доказательств высокого качества, так ранее проведенные исследования, в которых сравнивались исходы на основании данных о лучшем управлении назначением антимикробной терапии с выбранными клиническими или процессуальными исходами, можно отнести к категории «квази-исследования» [45, 103, 104]. Из всего этого можно сделать последовательный, но не универсальный, вывод о том, что все эти быстрые технологии по одиночке не способны привести даже к простому улучшению результатов в управлении антибактериальной терапией, не говоря уже об улучшении клинических исходов, без внедрения индивидуализированных (персонализированных) стратегий управления антибактериальной терапией, что отражено в руководящих принципах Американского общества инфекционных болезней (англ. the Infectious Diseases Society of America или IDSA) [45, 52, 101, 105, 106].
Целенаправленные стратегии управления антимикробной терапией, которые оценивались в целях поддержки быстрых диагностических тестов, привели к увеличению времени до момента принятия решения - уведомление члена группы управления антимикробной терапией, ожидание предоставления от него целевых прямых рекомендаций, другие действия, что увеличивают время взаимодействия клиницистов [105, 107]. Наиболее явный результат сочетания быстрых диагностических тестов с управлением антимикробной терапией - более оптимальное назначение антибиотиков и последующей де-эскалации, а вот такой показатель, как эффективность затрат, остается за кадром [45, 108, 109]. Влияние же на клинические исходы достаточно сильно различается от исследования к исследованию, в большинстве из них оцениваются длительность госпитализации, летальность и повторные госпитализации [110 - 112]. Причины таких расхождений тщательно не изучались, но, скорее всего, все это связано с опытом и экспертными знаниями применимости быстрых диагностических тестов [113, 114]. Вероятность проведения де-эскалации «в одночасье» в клинических условиях очень мала даже если команда, состоящая из микробиолога и специалистов по управлению за назначением антибиотиков, будет работать в режиме 24/7, когда с большей вероятностью в ночное время будут работать более неопытные специалисты, да и есть определенная связь с культурой оказания медицинской помощи (ухода) в ночное время [105].
Самые действенные результаты включают в себя (1) идентификация вероятной контаминации и (2) детекция молекулярных целей, коррелирующих с резистентностью, что препятствует назначению эмпирического режима. Одним из молекулярных быстрых диагностических тестов, что наиболее тщательно оценивал антимикробную чувствительность, тем самым оказывая клиническое влияние, стала система «the Verigene». Результаты для грамположительных микроорганизмов из посева крови, были явно более действенными, чем для грамотрицательных, что отражает сложность генетических маркеров резистентности, а также раннее субоптимальное лечение грамотрицательного сепсиса [105]. Мультиплексные ПЦР тесты ограничены количеством целевых генов, которые эти способны идентифицировать, что не покрывает потребности в определении всех релевантных механизмов резистентности.
Внедрение молекулярных быстрых диагностических тестов достаточно ресурсоемкая задача, при этом их внедрение не отменяет необходимости проведения фенотипического теста для определения антимикробной чувствительности [115]. К тому же молекулярные быстрые диагностические тесты имеют существенные ограничения в виде снижения чувствительности при детекции полимикробной бактериемии, при возможной кросс-контаминации или при идентичности генов (к примеру Shigella и E. coli), а также из-за ограниченного спектра механизмов резистентности и отсутствия клинически подтвержденной корреляции между генетическими маркерами и минимальной подавляющей концентрацией (англ. minimum inhibitory concentrations или MICs). Последнее может быть критичным для терапевтического лекарственного мониторинга и при наблюдении за «диким» типом патогена, а также и при определении возможности назначения меропенема при наличии карбапенемаз.
Принесет ли пользу внедрение быстрых диагностических тестов в ежедневную клиническую практику будет зависеть от самого лечебного учреждения, включая сюда и локальные антибиотикограммы, от тяжести пациентов, которым данное лечебное учреждение оказывает медицинскую помощь, от наличия программы по контролю за назначением антибиотиков и ее эффективности, от уровеня взаимодействия между микробиологами и интенсивистами (инфекционистами) [109]. Страны с низким и средним уровнем доходов, где высока распространенность микроорганизмов со множественной антибиотикорезистентностью, могут счесть стоимость молекулярных методов быстрой диагностики непомерно высокой, и здесь можно посоветовать применять фенотипические тесты или портативные методы с оптическим датчиком высокого разрешения [116].
Диагностика у кровати пациента (англ. Point of Care Diagnostics или РОС)
В настоящее время не существует систем для точной диагностики инфекций кровотока или большинства других критических инфекций непосредственно у кровати пациента (РоС). Есть некоторая надежда на будущие технологические достижения, такие как микрожидкостные устройства, которые могут интегрировать обработку проб и генерацию сигналов в рамках PоC, возможно, с использованием таких платформ, как иммуноанализ «на кристалле» или анализ нуклеиновых кислот. Теоретически такая технология может включать все ключевые этапы молекулярного обнаружения - лизис и экстракцию клеток, очистку нуклеиновых кислот, амплификацию и обнаружение продуктов реакции. Такая миниатюризация может также позволить мультиплексирование для обеспечения высокопроизводительного тестирования в одном портативном устройстве [118]. В настоящее нет готовых коммерческих продуктов для клинической оценки.
Выводы
Мы питаем надежду на то, что новые микробиологические методы увеличат наши
возможности в быстрой и точной идентификации патогенов у пациентов, находящихся
в критическом состоянии. Но все же необходимы исследования с хорошо продуманным
дизайном, в которых оценке подвергнутся ключевые клинические исходы, что
поможет нам определить роль новых микробиологических методов в улучшении
менеджмента тяжелых инфекций.

Список литературы
1. Vincent JL, Sakr Y, Sprung CL, Ranieri VM, Reinhart K, Gerlach H, et al. Sepsis in European intensive care units: results of the SOAP study. Crit Care Med. 2006;34(2):344–53.
2. Lagu T, Rothberg MB, Shieh MS, Pekow PS, Steingrub JS, Lindenauer PK. Hospitalizations, costs, and outcomes of severe sepsis in the United States 2003 to 2007. Crit Care Med. 2012;40(3):754–61.
3. Vincent JL, Rello J, Marshall J, Silva E, Anzueto A, Martin CD, et al. International study of the prevalence and outcomes of infection in intensive care units. JAMA. 2009;302(21):2323–9.
4. Denny KJ, De Waele J, Laupland KB, Harris PNA, Lipman J. When not to start antibiotics: avoiding antibiotic overuse in the intensive care unit. Clin Microbiol Infect. 2020;26(1):35–40.
5. Hall KK, Lyman JA. Updated review of blood culture contamination. Clin Microbiol Rev. 2006;19(4):788–802.
6. Murray PR, Masur H. Current approaches to the diagnosis of bacterial and fungal bloodstream infections in the intensive care unit. Crit Care Med. 2012;40(12):3277–82.
7. Rodriguez-Sanchez B, Cercenado E, Coste AT, Greub G. Review of the impact of MALDI-TOF MS in public health and hospital hygiene, 2018. Euro Surveill. 2019;24(4):1800193.
8. Messacar K, Parker SK, Todd JK, Dominguez SR. Implementation of rapid molecular infectious disease diagnostics: the role of diagnostic and antimicrobial stewardship. Journal of clinical microbiology. 2017;55(3):715–23.
9. Trotter AJ, Aydin A, Strinden MJ, O'Grady J. Recent and emerging technologies for the rapid diagnosis of infection and antimicrobial resistance. Curr Opin Microbiol. 2019;51:39–45.
10. Minassian AM, Newnham R, Kalimeris E, Bejon P, Atkins BL, Bowler IC. Use of an automated blood culture system (BD BACTEC) for diagnosis of prosthetic joint infections: easy and fast. BMC Infect Dis. 2014;14:233.
11. Li Z, Liu S, Chen H, Zhang X, Ling Y, Zhang N, et al. Comparative evaluation of BACTEC FX, BacT/ALERT 3D, and BacT/ALERT VIRTUO-automated blood culture systems using simulated blood cultures. Acta Clin Belg. 2020:1–8.
12. Menchinelli G, Liotti FM, Fiori B, De Angelis G, D'Inzeo T, Giordano L, et al. In vitro Evaluation of BACT/ALERT(R) VIRTUO(R), BACT/ALERT 3D(R), and BACTEC FX automated blood culture systems for detection of microbial pathogens using simulated human blood samples. Front Microbiol. 2019;10:221.
13. Chung Y, Kim IH, Han M, Kim HS, Kim HS, Song W, et al. A comparative evaluation of BACT/ALERT FA PLUS and FN PLUS blood culture bottles and BD BACTEC Plus Aerobic and Anaerobic blood culture bottles for antimicrobial neutralization. Eur J Clin Microbiol Infect Dis. 2019;38(12):2229–33.
14. Somily AM, Habib HA, Torchyan AA, Sayyed SB, Absar M, AlAqeel R, et al. Time-to-detection of bacteria and yeast with the BACTEC FX versus BacT/Alert Virtuo blood culture systems. Ann Saudi Med. 2018;38(3):194–9.
15. Park J, Han S, Shin S. Comparison of growth performance of the BacT/ALERT VIRTUO and BACTEC FX Blood Culture Systems under simulated bloodstream infection conditions. Clin Lab. 2017;63(1):39–46.
16. Chen IH, Nicolau DP, Kuti JL. Effect of clinically meaningful antibiotic concentrations on recovery of Escherichia coli and Klebsiella pneumoniae isolates from anaerobic blood culture bottles with and without antibiotic binding resins. J Clin Microbiol. 2019;57(12).
17. Chen IH, Nicolau DP, Kuti JL. Recovery of Gram-negative bacteria from aerobic blood culture bottles containing antibiotic binding resins after exposure to beta-lactam and fluoroquinolone concentrations. J Clin Microbiol. 2019;57(10).
18. Wilson ML, Weinstein MP, Reller LB. Automated blood culture systems. Clin Lab Med. 1994;14(1):149–69.
19. Edmiston CE, Garcia R, Barnden M, DeBaun B, Johnson HB. Rapid diagnostics for bloodstream infections: a primer for infection preventionists. American journal of infection control. 2018;46(9):1060–8.
20. Smith KP, Kang AD, Kirby JE. Automated interpretation of blood culture Gram stains by use of a deep convolutional neural network. J Clin Microbiol. 2018;56(3).
21. Qian Q, Eichelberger K, Kirby JE. Rapid identification of Staphylococcus aureus in blood cultures by use of the direct tube coagulase test. J Clin Microbiol. 2007;45(7):2267–9.
22. Ozen NS, Ogunc D, Mutlu D, Ongut G, Baysan BO, Gunseren F. Comparison of four methods for rapid identification of Staphylococcus aureus directly from BACTEC 9240 blood culture system. Indian J Med Microbiol. 2011;29(1):42–6.
23. Chandrasekaran S, Abbott A, Campeau S, Zimmer BL, Weinstein M, Thrupp L, et al. Direct-from-blood-culture disk diffusion to determine antimicrobial susceptibility of Gram-negative bacteria: preliminary report from the Clinical and Laboratory Standards Institute Methods Development and Standardization Working Group. J Clin Microbiol. 2018;56(3).
24. Jonasson E, Matuschek E, Kahlmeter G. The EUCAST rapid disc diffusion method for antimicrobial susceptibility testing directly from positive blood culture bottles. J Antimicrob Chemother. 2020;75(4):968–78.
25. Kumar M, Shergill SPS, Tandel K, Sahai K, Gupta RM. Direct antimicrobial susceptibility testing from positive blood culture bottles in laboratories lacking automated antimicrobial susceptibility testing systems. Med J Armed Forces India. 2019;75(4): 450–7.
26. Bayraktar B, Baris A, Malkocoglu G, Erdemir D, Kina N. Comparison of Carba NP-direct, carbapenem inactivation method, and beta-CARBA tests for detection of carbapenemase production in Enterobacteriaceae. Microb Drug Resist. 2019;25(1): 97–102.
27. Cunningham SA, Limbago B, Traczewski M, Anderson K, Hackel M, Hindler J, et al. Multicenter performance assessment of Carba NP Test. J Clin Microbiol. 2017;55(6):1954–60.
28. McMullen AR, Wallace MA, LaBombardi V, Hindler J, Campeau S, Humphries R, et al. Multicenter evaluation of the RAPIDEC(R) CARBA NP assay for the detection of carbapenemase production in clinical isolates of Enterobacterales and Pseudomonas aeruginosa. Eur J Clin Microbiol Infect Dis. 2020;39(11):2037–44.
29. Blanc DS, Poncet F, Grandbastien B, Greub G, Senn L, Nordmann P. Evaluation of the performance of rapid tests for screening carriers of acquired ESBL producers and their impact on the turnaround time. J Hosp Infect. 2020.
30. Couturier MR, Graf EH, Griffin AT. Urine antigen tests for the diagnosis of respiratory infections: legionellosis, histoplasmosis, pneumococcal pneumonia. Clin Lab Med. 2014;34(2):219–36.
31. Viasus D, Calatayud L, McBrown MV, Ardanuy C, Carratala J. Urinary antigen testing in community-acquired pneumonia in adults: an update. Expert Rev Anti Infect Ther. 2019;17(2):107–15.
32. Avni T, Bieber A, Green H, Steinmetz T, Leibovici L, Paul M. Diagnostic accuracy of PCR alone and compared to urinary antigen testing for detection of Legionella spp.: a systematic review. J Clin Microbiol. 2016;54(2):401–11.
33. Baggett HC, Rhodes J, Dejsirilert S, Salika P, Wansom T, Jorakate P, et al. Pneumococcal antigen testing of blood culture broth to enhance the detection of Streptococcus pneumoniae bacteremia. Eur J Clin Microbiol Infect Dis. 2012;31(5):753–6.
34. Kassis C, Zaidi S, Kuberski T, Moran A, Gonzalez O, Hussain S, et al. Role of Coccidioides antigen testing in the cerebrospinal fluid for the diagnosis of coccidioidal meningitis. Clin Infect Dis. 2015;61(10):1521–6.
35. Porcel JM. Biomarkers in the diagnosis of pleural diseases: a 2018 update. Ther Adv Respir Dis. 2018;12:1753466618808660.
36. Perkins MD, Mirrett S, Reller LB. Rapid bacterial antigen detection is not clinically useful. J Clin Microbiol. 1995;33(6):1486–91.
37. Andrews D, Chetty Y, Cooper BS, Virk M, Glass SK, Letters A, et al. Multiplex PCR point of care testing versus routine, laboratory-based testing in the treatment of adults with respiratory tract infections: a quasi-randomised study assessing impact on length of stay and antimicrobial use. BMC Infect Dis. 2017;17(1):671.
38. Yang S, Rothman RE. PCR-based diagnostics for infectious diseases: uses, limitations, and future applications in acute-care settings. Lancet Infect Dis. 2004;4(6):337–48.
39. Pierrakos C, Vincent JL. Sepsis biomarkers: a review. Crit Care. 2010;14(1):R15.
40. Cui N, Zhang H, Chen Z, Yu Z. Prognostic significance of PCT and CRP evaluation for adult ICU patients with sepsis and septic shock: retrospective analysis of 59 cases. J Int Med Res. 2019;47(4):1573–9.
41. Van den Bruel A, Thompson MJ, Haj-Hassan T, Stevens R, Moll H, Lakhanpaul M, et al. Diagnostic value of laboratory tests in identifying serious infections in febrile children: systematic review. BMJ. 2011;342:d3082.
42. Grondman I, Pirvu A, Riza A, Ioana M, Netea MG. Biomarkers of inflammation and the etiology of sepsis. Biochem Soc Trans. 2020;48(1):1–14.
43. de Jong E, van Oers JA, Beishuizen A, Vos P, Vermeijden WJ, Haas LE, et al. Efficacy and safety of procalcitonin guidance in reducing the duration of antibiotic treatment in critically ill patients: a randomised, controlled, open-label trial. Lancet Infect Dis. 2016;16(7):819–27.
44. Stocker M, van Herk W, El Helou S, Dutta S, Fontana MS, Schuerman F, et al. Procalcitonin-guided decision making for duration of antibiotic therapy in neonates with suspected early-onset sepsis: a multicentre, randomised controlled trial (NeoPIns). Lancet. 2017;390(10097):871–81.
45. Timbrook TT, Morton JB, McConeghy KW, Caffrey AR, Mylonakis E, LaPlante KL. The effect of molecular rapid diagnostic testing on clinical outcomes in bloodstream infections: a systematic review and meta-analysis. Clin Infect Dis. 2017;64(1):15–23.
46. Tansarli GS, Chapin KC. Diagnostic test accuracy of the BioFire(R) FilmArray(R) meningitis/encephalitis panel: a systematic review and meta-analysis. Clin Microbiol Infect. 2020;26(3): 281–90.
47. Dack K, Pankow S, Ablah E, Zackula R, Assi M. Contribution of the BioFire((R)) FilmArray((R)) meningitis/encephalitis panel: assessing antimicrobial duration and length of stay. Kans J Med. 2019;12(1):1–3.
48. Moffa MA, Bremmer DN, Carr D, Buchanan C, Shively NR, Elrufay R, et al. Impact of a multiplex polymerase chain reaction assay on the clinical management of adults undergoing a lumbar puncture for suspected community-onset central nervous system infections. Antibiotics (Basel). 2020;9(6).
49. Maataoui N, Chemali L, Patrier J, Tran Dinh A, Le Fevre L, LortatJacob B, et al. Impact of rapid multiplex PCR on management of antibiotic therapy in COVID-19-positive patients hospitalized in intensive care unit. Eur J Clin Microbiol Infect Dis. 2021.
50. Vandendriessche S, Padalko E, Wollants E, Verfaillie C, Verhasselt B, Coorevits L. Evaluation of the Seegene Allplex Respiratory Panel for diagnosis of acute respiratory tract infections. Acta Clin Belg. 2019;74(6):379–85.
51. Blaschke AJ, Heyrend C, Byington CL, Fisher MA, Barker E, Garrone NF, et al. Rapid identification of pathogens from positive blood cultures by multiplex polymerase chain reaction using the FilmArray system. Diagn Microbiol Infect Dis. 2012;74(4):349–55.
52. Southern TR, VanSchooneveld TC, Bannister DL, Brown TL, Crismon AS, Buss SN, et al. Implementation and performance of the BioFire FilmArray(R) Blood Culture Identification panel with antimicrobial treatment recommendations for bloodstream infections at a midwestern academic tertiary hospital. Diagn Microbiol Infect Dis. 2015;81(2):96–101.
53. Verroken A, Despas N, Rodriguez-Villalobos H, Laterre PF. The impact of a rapid molecular identification test on positive blood cultures from critically ill with bacteremia: a pre-post intervention study. PLoS One. 2019;14(9):e0223122.
54. Sansot M, Fradin E, Chenouard R, Kempf M, Kouatchet A, Lasocki S, et al. Performance of the extended use of the FilmArray((R)) BCID panel kit for bronchoalveolar lavage analysis. Mol Biol Rep. 2019;46(3):2685–92.
55. Spencer DH, Sellenriek P, Burnham CA. Validation and implementation of the GeneXpert MRSA/SA blood culture assay in a pediatric setting. Am J Clin Pathol. 2011;136(5):690–4.
56. Cointe A, Walewski V, Hobson CA, Doit C, Bidet P, Dortet L, et al. Rapid carbapenemase detection with Xpert Carba-R V2 directly on positive blood vials. Infect Drug Resist. 2019;12:3311–6.
57. Mancini N, Infurnari L, Ghidoli N, Valzano G, Clementi N, Burioni R, et al. Potential impact of a microarray-based nucleic acid assay for rapid detection of Gram-negative bacteria and resistance markers in positive blood cultures. J Clin Microbiol. 2014;52(4):1242–5.
58. Buchan BW, Ginocchio CC, Manii R, Cavagnolo R, Pancholi P, Swyers L, et al. Multiplex identification of gram-positive bacteria and resistance determinants directly from positive blood culture broths: evaluation of an automated microarray-based nucleic acid test. PLoS Med. 2013;10(7):e1001478.
59. Evans SR, Tran TTT, Hujer AM, Hill CB, Hujer KM, Mediavilla JR, et al. Rapid molecular diagnostics to inform empiric use of ceftazidime/avibactam and ceftolozane/tazobactam against Pseudomonas aeruginosa: PRIMERS IV. Clin Infect Dis. 2019;68(11):1823–30.
60. Claeys KC, Heil EL, Hitchcock S, Johnson JK, Leekha S. Management of Gram-negative bloodstream infections in the era of rapid diagnostic testing: impact with and without antibiotic stewardship. Open Forum Infect Dis. 2020;7(10):ofaa427.
61. Tribble AC, Gerber JS, Bilker WB, Lautenbach E. Impact of rapid diagnostics with antimicrobial stewardship support for children with positive blood cultures: a quasi-experimental study with time trend analysis. Infect Control Hosp Epidemiol. 2020;41(8):883–90.
62. Laub RR, Knudsen JD. Clinical consequences of using PNAFISH in Staphylococcal bacteraemia. Eur J Clin Microbiol Infect Dis. 2014;33(4):599–601.
63. Cosgrove SE, Li DX, Tamma PD, Avdic E, Hadhazy E, Wakefield T, et al. Use of PNA FISH for blood cultures growing Gram-positive cocci in chains without a concomitant antibiotic stewardship intervention does not improve time to appropriate antibiotic therapy. Diagn Microbiol Infect Dis. 2016;86(1):86–92.
64. Stevenson M, Pandor A, Martyn-St James M, Rafia R, Uttley L, Stevens J, et al. Sepsis: the LightCycler SeptiFast Test MGRADE(R), SepsiTest and IRIDICA BAC BSI assay for rapidly identifying bloodstream bacteria and fungi - a systematic review and economic evaluation. Health Technol Assess. 2016;20(46):1–246.
65. Dark P, Blackwood B, Gates S, McAuley D, Perkins GD, McMullan R, et al. Accuracy of LightCycler((R)) SeptiFast for the detection and identification of pathogens in the blood of patients with suspected sepsis: a systematic review and meta-analysis. Intensive Care Med. 2015;41(1):21–33.
66. Chang SS, Hsieh WH, Liu TS, Lee SH, Wang CH, Chou HC, et al. Multiplex PCR system for rapid detection of pathogens in patients with presumed sepsis - a systemic review and meta-analysis. PLoS One. 2013;8(5):e62323.
67. Ziegler I, Fagerstrom A, Stralin K, Molling P. Evaluation of a commercial multiplex PCR assay for detection of pathogen DNA in blood from patients with suspected sepsis. PLoS One. 2016;11(12):e0167883.
68. Zboromyrska Y, Cilloniz C, Cobos-Trigueros N, Almela M, Hurtado JC, Vergara A, et al. Evaluation of the Magicplex Sepsis real-time test for the rapid diagnosis of bloodstream infections in adults. Front Cell Infect Microbiol. 2019;9:56.
69. Pfaller MA, Wolk DM, Lowery TJ. T2MR and T2Candida: novel technology for the rapid diagnosis of candidemia and invasive candidiasis. Future Microbiol. 2016;11(1):103–17.
70. Mylonakis E, Clancy CJ, Ostrosky-Zeichner L, Garey KW, Alangaden GJ, Vazquez JA, et al. T2 magnetic resonance assay for the rapid diagnosis of candidemia in whole blood: a clinical trial. Clin Infect Dis. 2015;60(6):892–9.
71. Clancy CJ, Nguyen MH. T2 magnetic resonance for the diagnosis of bloodstream infections: charting a path forward. J Antimicrob Chemother. 2018;73(suppl_4):iv2–5.
72. De Angelis G, Posteraro B, De Carolis E, Menchinelli G, Franceschi F, Tumbarello M, et al. T2Bacteria magnetic resonance assay for the rapid detection of ESKAPEc pathogens directly in whole blood. J Antimicrob Chemother. 2018;73(suppl_4):iv20–iv6.
73. Nguyen MH, Clancy CJ, Pasculle AW, Pappas PG, Alangaden G, Pankey GA, et al. Performance of the T2Bacteria panel for diagnosing bloodstream infections: a diagnostic accuracy study. Ann Intern Med. 2019;170(12):845–52.
74. Maki DG. The T2Bacteria Panel had 90% sensitivity for detecting targeted organisms, 43% for any bloodstream infection organism. Ann Intern Med. 2019;171(6):JC34.
75. Weinrib DA, Capraro GA. The uncertain clinical benefit of the T2Bacteria panel. Ann Intern Med. 2019;170(12):888–9.
76. Oros D, Ceprnja M, Zucko J, Cindric M, Hozic A, Skrlin J, et al. Identification of pathogens from native urine samples by MALDITOF/TOF tandem mass spectrometry. Clin Proteomics. 2020;17:25.
77. Brunetti G, Ceccarelli G, Giordano A, Navazio AS, Vittozzi P, Venditti M, et al. Fast and reliable diagnosis of XDR Acinetobacter baumannii meningitis by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. New Microbiol. 2018;41(1):77–9.
78. Ozenci V, Patel R, Ullberg M, Stralin K. Demise of polymerase chain reaction/electrospray ionization-mass spectrometry as an infectious diseases diagnostic tool. Clin Infect Dis. 2018;66(3):452–5.
79. Rutanga JP, Van Puyvelde S, Heroes AS, Muvunyi CM, Jacobs J, Deborggraeve S. 16S metagenomics for diagnosis of bloodstream infections: opportunities and pitfalls. Expert Rev Mol Diagn. 2018;18(8):749–59.
80. Chiu CY, Miller SA. Clinical metagenomics. Nat Rev Genet. 2019;20(6):341–55.
81. Parize P, Muth E, Richaud C, Gratigny M, Pilmis B, Lamamy A, et al. Untargeted next-generation sequencing-based first-line diagnosis of infection in immunocompromised adults: a multicentre, blinded, prospective study. Clin Microbiol Infect. 2017;23(8):574 e1–6.
82. Blauwkamp TA, Thair S, Rosen MJ, Blair L, Lindner MS, Vilfan ID, et al. Analytical and clinical validation of a microbial cell-free DNA sequencing test for infectious disease. Nat Microbiol. 2019;4(4):663–74.
83. Goggin KP, Gonzalez-Pena V, Inaba Y, Allison KJ, Hong DK, Ahmed AA, et al. Evaluation of plasma microbial cell-free DNA sequencing to predict bloodstream infection in pediatric patients with relapsed or refractory cancer. JAMA Oncol. 2020;6(4):552–6.
84. Niles DT, Wijetunge DSS, Palazzi DL, Singh IR, Revell PA. Plasma metagenomic next-generation sequencing assay for identifying pathogens: a retrospective review of test utilization in a large children’s hospital. J Clin Microbiol. 2020;58(11).
85. Hogan CA, Yang S, Garner OB, Green DA, Gomez CA, Dien Bard J, et al. Clinical impact of metagenomic next-generation sequencing of plasma cell-free DNA for the diagnosis of infectious diseases: a multicenter retrospective cohort study. Clin Infect Dis. 2020.
86. Grumaz S, Grumaz C, Vainshtein Y, Stevens P, Glanz K, Decker SO, et al. Enhanced performance of next-generation sequencing diagnostics compared with standard of care microbiological diagnostics in patients suffering from septic shock. Crit Care Med. 2019;47(5):e394–402.
87. Charnot-Katsikas A, Tesic V, Love N, Hill B, Bethel C, Boonlayangoor S, et al. Use of the Accelerate Pheno system for identification and antimicrobial susceptibility testing of pathogens in positive blood cultures and impact on time to results and workflow. J Clin Microbiol. 2018;56(1).
88. Lutgring JD, Bittencourt C, McElvania TeKippe E, Cavuoti D, Hollaway R, Burd EM. Evaluation of the Accelerate Pheno System: results from two academic medical centers. J Clin Microbiol. 2018;56(4).
89. Marschal M, Bachmaier J, Autenrieth I, Oberhettinger P, Willmann M, Peter S. Evaluation of the Accelerate Pheno System for fast identification and antimicrobial susceptibility testing from positive blood cultures in bloodstream infections caused by Gram-negative pathogens. J Clin Microbiol. 2017;55(7):2116–26.
90. Pancholi P, Carroll KC, Buchan BW, Chan RC, Dhiman N, Ford B, et al. Multicenter evaluation of the Accelerate PhenoTest BC kit for rapid identification and phenotypic antimicrobial susceptibility testing using morphokinetic cellular analysis. J Clin Microbiol. 2018;56(4).
91. Dubourg G, Raoult D. Emerging methodologies for pathogen identification in positive blood culture testing. Expert Rev Mol Diagn. 2016;16(1):97–111.
92. Lee M, Scardina T, Zheng X, Patel SJ. Clinical performance and impact of Accelerate Pheno for Gram-negative bacteremia in hospitalized children. Clin Ther. 2020;42(9):1630–6.
93. Dare RK, Lusardi K, Pearson C, McCain KD, Daniels B, Van S, et al. Clinical impact of Accelerate PhenoTM rapid blood culture detection system in bacteremic patients. Clin Infect Dis. 2020.
94. Vasala A, Hytonen VP, Laitinen OH. Modern tools for rapid diagnostics of antimicrobial resistance. Front Cell Infect Microbiol. 2020;10:308.
95. Hsueh EC, DeBloom JR, Lee J, Sussman JJ, Covington KR, Middlebrook B, et al. Interim analysis of survival in a prospective, multi-center registry cohort of cutaneous melanoma tested with a prognostic 31-gene expression profile test. J Hematol Oncol. 2017;10(1):152.
96. Vargas J, Lima JA, Kraus WE, Douglas PS, Rosenberg S. Use of the Corus(R) CAD Gene expression test for assessment of obstructive coronary artery disease likelihood in symptomatic nondiabetic patients. PLoS Curr. 2013;5: ecurrents.eogt.0f04f6081905998fa92b99593478aeab.
97. Sweeney TE, Wong HR, Khatri P. Robust classification of bacterial and viral infections via integrated host gene expression diagnostics. Sci Transl Med. 2016;8(346):346ra91.
98. Herberg JA, Kaforou M, Wright VJ, Shailes H, Eleftherohorinou H, Hoggart CJ, et al. Diagnostic test accuracy of a 2-transcript host RNA signature for discriminating bacterial vs viral infection in febrile children. JAMA. 2016;316(8):835–45.
99. Sampson D, Yager TD, Fox B, Shallcross L, McHugh L, Seldon T, et al. Blood transcriptomic discrimination of bacterial and viral infections in the emergency department: a multi-cohort observational validation study. BMC Med. 2020;18(1):185.
100. Wong HR, Cvijanovich NZ, Anas N, Allen GL, Thomas NJ, Bigham MT, et al. Endotype transitions during the acute phase of pediatric septic shock reflect changing risk and treatment response. Crit Care Med. 2018;46(3):e242–e9.
101. Palavecino EL, Williamson JC, Ohl CA. Collaborative antimicrobial stewardship: working with microbiology. Infect Dis Clin North Am. 2020;34(1):51–65.
102. Aamot HV, Noone JC, Skramm I, Leegaard TM. Are conventional microbiological diagnostics sufficiently expedient in the era of rapid diagnostics? Evaluation of conventional microbiological diagnostics of orthopedic implant-associated infections (OIAI). Acta Orthop. 2020:1–6.
103. Perez KK, Olsen RJ, Musick WL, Cernoch PL, Davis JR, Land GA, et al. Integrating rapid pathogen identification and antimicrobial stewardship significantly decreases hospital costs. Arch Pathol Lab Med. 2013;137(9):1247–54.
104. Huang AM, Newton D, Kunapuli A, Gandhi TN, Washer LL, Isip J, et al. Impact of rapid organism identification via matrix-assisted laser desorption/ionization time-of-flight combined with antimicrobial stewardship team intervention in adult patients with bacteremia and candidemia. Clin Infect Dis. 2013;57(9):1237–45.
105. Juttukonda LJ, Katz S, Gillon J, Schmitz J, Banerjee R. Impact of a rapid blood culture diagnostic test in a children’s hospital depends on Gram-positive versus Gram-negative organism and day versus night shift. J Clin Microbiol. 2020;58(4).
106. Banerjee R, Teng CB, Cunningham SA, Ihde SM, Steckelberg JM, Moriarty JP, et al. Randomized trial of rapid multiplex polymerase chain reaction-based blood culture identification and susceptibility testing. Clin Infect Dis. 2015;61(7):1071–80.
107. Cwengros LN, Mynatt RP, Timbrook TT, Mitchell R, Salimnia H, Lephart P, et al. Minimizing time to optimal antimicrobial therapy for Enterobacteriaceae bloodstream infections: a retrospective, hypothetical application of predictive scoring tools vs rapid diagnostics tests. Open Forum Infect Dis. 2020;7(8):ofaa278.
108. Kondo M, Simon MS, Westblade LF, Jenkins SG, Babady NE, Loo AS, et al. Implementation of infectious diseases rapid molecular diagnostic tests and antimicrobial stewardship program involvement in acute-care hospitals. Infect Control Hosp Epidemiol. 2020:1–3.
109. She RC, Bender JM. Advances in rapid molecular blood culture diagnostics: healthcare impact, laboratory implications, and multiplex technologies. J Appl Lab Med. 2019;3(4):617–30.
110. Nasef R, El Lababidi R, Alatoom A, Krishnaprasad S, Bonilla F. The impact of integrating rapid PCR-based blood culture identification panel to an established antimicrobial stewardship program in the United Arab of Emirates. Int J Infect Dis. 2020;91:124–8.
111. Mahrous AJ, Thabit AK, Elarabi S, Fleisher J. Clinical impact of pharmacist-directed antimicrobial stewardship guidance following blood culture rapid diagnostic testing. J Hosp Infect. 2020;106(3): 436–46.
112. Hogan CA, Ebunji B, Watz N, Kapphahn K, Rigdon J, Mui E, et al. Impact of rapid antimicrobial susceptibility testing in Gram-negative rod bacteremia: a quasi-experimental study. J Clin Microbiol. 2020;58(9).
113. Faugno AK, Laidman AY, Perez Martinez JD, Campbell AJ, Blyth CC. Do rapid diagnostic methods improve antibiotic prescribing in paediatric bacteraemia? J Paediatr Child Health. 2020.
114. Tseng AS, Kasule SN, Rice F, Mi L, Chan L, Seville MT, et al. Is It Actionable? An evaluation of the rapid PCR-based blood culture identification panel on the management of Gram-positive and Gram-negative blood stream infections. Open Forum Infect Dis. 2018;5(12):ofy308.
115. Oberhettinger P, Zieger J, Autenrieth I, Marschal M, Peter S. Evaluation of two rapid molecular test systems to establish an algorithm for fast identification of bacterial pathogens from positive blood cultures. Eur J Clin Microbiol Infect Dis. 2020;39(6): 1147–57.
116. Kapur S, Gehani M, Kammili N, Bhardwaj P, Nag V, Devara SM, et al. Clinical validation of innovative optical-sensor-based, low-cost, rapid diagnostic test to reduce antimicrobial resistance. J Clin Med. 2019;8(12).
117. National Pathology Accreditation Advisory Council (NPAAC). Guidelines for point of care testing. In: Department of Health, editor. 1st Edition ed 2015.
118. Kozel TR, Burnham-Marusich AR. Point-of-care testing for infectious diseases: past, present, and future. J Clin Microbiol. 2017;55(8):2313–20.
Current Infectious Disease Reports (2021) 23:12